A nagy genetikai őskeresés

Két év, nyolc hónap, huszonnyolc éjszaka. Salman Rushdie Ezeregyéjszaka-ihlette, mágikus realizmussal átszőtt legutóbbi regényében ennyi időre van szükség, amíg ibn Rusd, a középkori bölcs és Dunja, a dzsinn hercegnő jelenkori utódjai felülkerekednek a Földet megtámadó gonosz dzsinnseregeken, a fizika törvényei pedig visszazökkenhetnek a normális kerékvágásba. A történet szerint az évszázadokkal ezelőtti különös frigy ma is élő leszármazottai paranormális képességeik mellett elsősorban cimpátlan fülük miatt tűnnek ki a népességből – egyszersmind azt is sugallva, hogy a két különleges tulajdonság együttesen száll az utódokra. Rushdie célja aligha az öröklődéssel kapcsolatos ismeretterjesztés volt. A mű értékéből így nem von le, hogy e nem túl lényeges elem tökéletes tükre annak, hogy a közbeszédben miféle naiv és pontatlan kép él az öröklődésről.

A „tiszta anyja/apja ez a gyerek” és a „génjeiben van” frázisok mögött több az előítélet, mint a tudomány. Mindez részben azzal is magyarázható, hogy az öröklődéstan (köz)oktatása szinte egyáltalán nem tartott lépést az elmúlt bő évtized felfedezéseivel, így ez a vulgárgenetika elavult, meghaladott gondolkodásmódot képvisel.
A 20. század egyik legnagyobb felismerése az volt, hogy a tulajdonságok öröklődéséért a korábban unalmasnak gondolt dezoxiribonukleinsav, közismerten DNS-molekula a felelős. Ez a mindössze négy nukleotid váltakozásából létrejövő molekula egy jól meghatározott, az élővilágban közel univerzális módon kódolja a sejt legtöbb funkcióját és jellegét meghatározó fehérjéket. A hogyan fogós kérdés. Olyannyira, hogy elmúlt fél évszázad központi kérdése a biológiában, hogy milyen módon szabályozható a gének működése: ha minden sejtünk ugyanazt a DNS-t hordozza, azok miért lesznek mégis különbözőek?
Csavarodik, pöndörödik
Egy átlagos emberi sejt durván 6 milliárd bázispárnyi DNS-t hordoz: ennek felét az apai, másik felét pedig az anyai ivarsejtek hozzák, összeolvadásukat követően így jön létre az embrió. Ez persze egyszersmind azt is jelenti, hogy testi sejtjeink a legtöbb génünkből két kópiát (allélt) hordoznak, (szakszóval: diploidok). A két allél nagyon gyakran nem pont azonos. A különbség lehet egészen minimális, ami a DNS szekvenciájában felismerhető ugyan, de nem okoz működésbeli különbséget a két allél terméke között, de az is előfordul, hogy az egyik allél egyáltalán nem működik, a másik viszont igen. De az is, hogy mindkét allél működésképtelen és ilyenkor egy genetikai gyökerű betegséggel állunk szemben.1 (A tankönyvek kedvelt példája, a sarlósejtes vérszegénység tipikusan ilyen.)
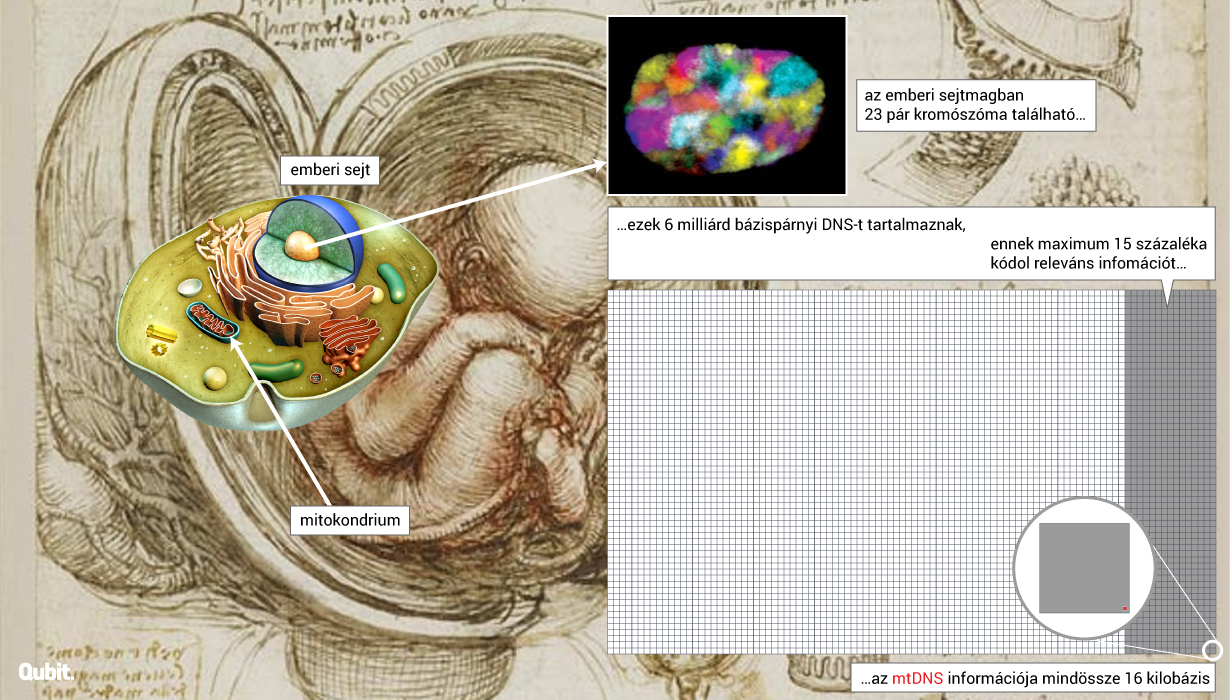
Az egy-egy emberi sejtben fellelhető 6 milliárd bázis 23 pár (vagyis összesen 46 darab) folytonos egységet, azaz kromoszómát hoz létre. Ezek közül a legnagyobb durván 250 millió bázispárból, míg a legkisebb alig 50 millióból áll. Egy átlagos emberi sejt magjában a kromoszómák rendezetlen gombolyagot alkotnak, és csak a sejtek osztódásakor, a DNS megkettőződése után jelennek meg – a kis túlzással popkult-ikonná avanzsáló – klasszikus X-alakú kromoszómák. Ha az osztódás ivarsejtek kialakulását eredményezi, akkor számfelező osztódásról (meiózisról) beszélünk: a kialakuló sejtekben 23 pár kromoszóma helyett már csak 23 darab található. Hogy ne legyen ilyen egyszerű, az osztódás kezdeti szakasza megkeveri a szülői genetikai állományokat, és az apai és anyai homológ kromoszómák nagy (több millió bázispárt kitevő) DNS-blokkokat cserélnek ki, miszerint rekombinálódnak. A folyamat eredményeként új DNS-kombinációt tartalmazó kromoszómák alakulnak ki – szakmai vélemények szerint konkrétan ez a szex (biológiai) lényege és evolúciós célja, egyszersmind értelme.2

Ebből azonban az is következik, hogy minél távolabbi egy ősünk, annál kevesebb DNS-t örököltünk tőle. Már nagyszüleink között is találhatunk olyat, akinek egy-egy kromoszómájából már nem hordozunk semmit, az időben hét generációt visszamenve pedig már olyan ősökre lelhetünk, akiktől semmiféle DNS-beli információt nem örököltünk. És ahogy hátrálunk a múltba, mindez egyre gyakoribb, olyannyira, hogy bő ezer évvel ezelőttről ugyan sok millió őst tudnánk beazonosítani egy részletes családfán, (erre majd még visszatérünk), ám DNS-t is örökítő genetikai ősből nagyjából mindössze 2600-at.
Ez nem azt jelenti, hogy a felmenők, akiknek genetikai anyagát nem hordozom, nem is az őseim. Nélkülük értelemszerűen nem lennék én sem. De a hozzám vezető leszármazási fa meiózisainak genetikai „gyufahúzásán” az ő genetikai információjuk a rövidebbet húzta, és elveszett. (Hogy még kuszább legyen: ettől másoknak még lehetnek ők a genetikai ősei.)
Mi fán terem a matrilinearitás?
Két olyan rövidebb DNS-darab van a sejtben, ami nem a fenti szabály szerint öröklődik, mert nincs alkalma a genetikai információt szétziláló rekombinációra. Az egyik az Y kromoszóma, a másik az úgynevezett mitokondriális DNS (mtDNS). Előbbi egy olyan kulcsgént, az SRY-t hordozza, amely – ahogy azt hivatalosan mondják – szükséges és elégséges feltétele, hogy egy egyed hím legyen.3 Normál esetben egyetlen Y kromoszóma van egy sejtben, értelemszerűen a rajta levő genetikai információ legnagyobb része osztódáskor nem is tud párba állni egy másik kromoszómával – márpedig enélkül nincs rekombináció sem.4 Így az Y kromoszóma, a DNS-ének másolásakor kialakuló viszonylag ritka mutációktól eltekintve, változatlanul száll apáról fiúra, sok-sok generáción át. (A családfa apai ágait figyelembe vevő leszármazási vonalat nevezzük patrilineárisnak.)
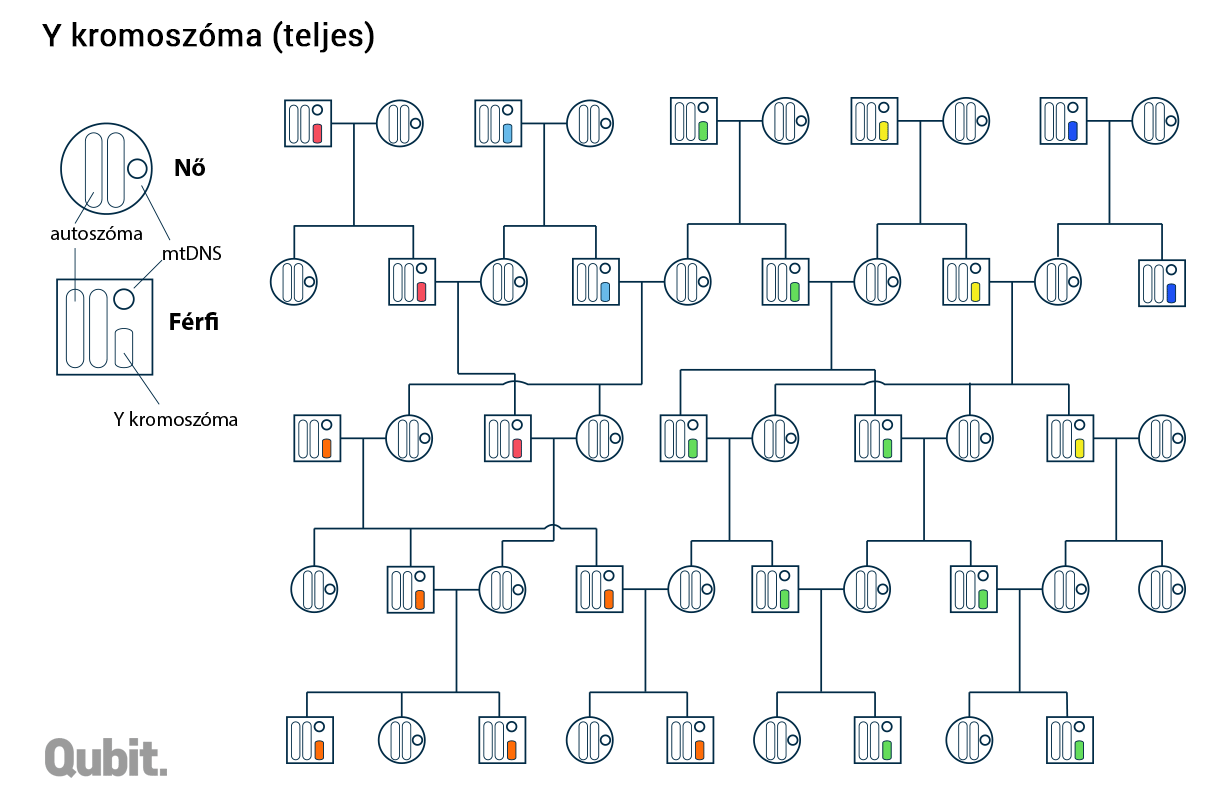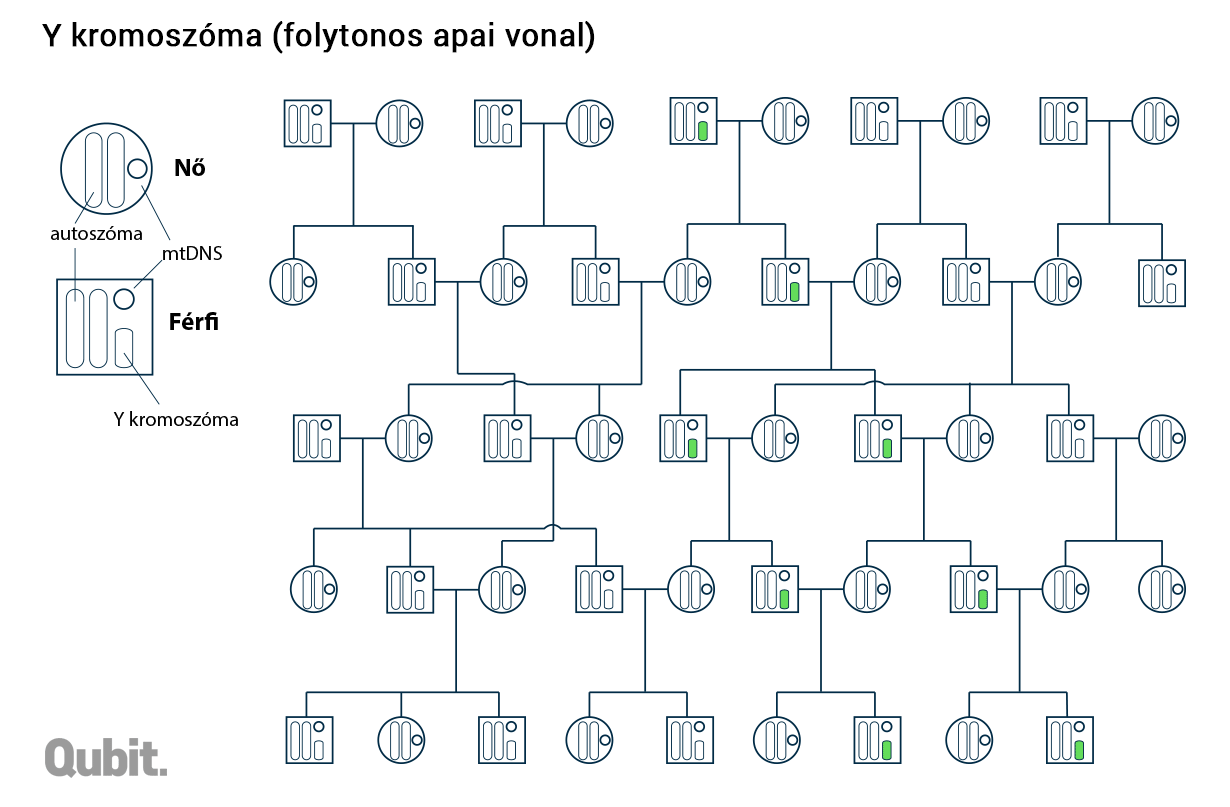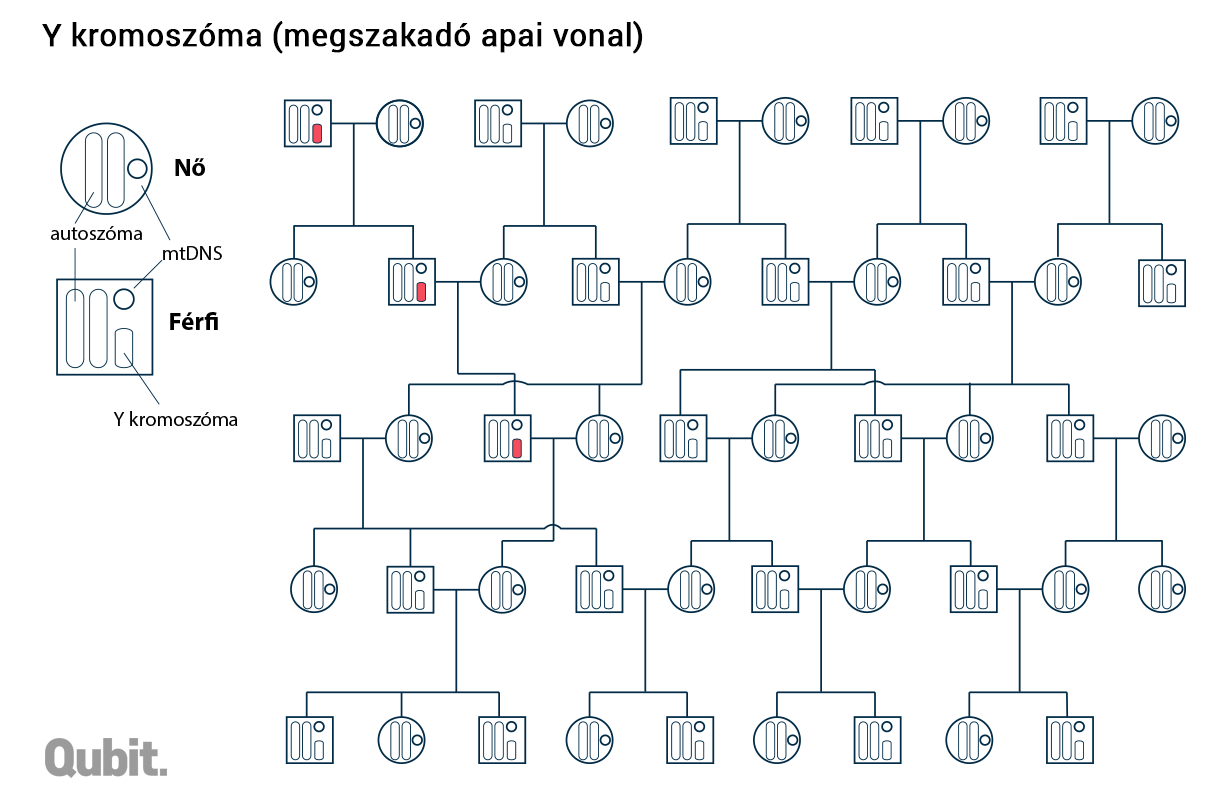
Az Y kromoszóma öröklődésének tükörképe az mtDNS-é, a különbség csak annyi, hogy az utóbbi a matrilineáris vagyis anyai vonalon adódik tovább. Ez a rövid (alig 16 ezer bázisnyi), gyűrű alakú DNS-molekula, ahogy azt a neve is sugallja, a sejtek energiatermelését végző mitokondriumokban lelhető fel, és öröklődése teljesen más logikát követ, mint a sejtmagban levő örökítőanyagé: a mitokondriumok osztódással szaporodnak, és eközben lemásolódik a bennük levő mtDNS is. Érdekes, hogy a leendő embrió mtDNS-e kizárólag a petesejtből származik (vagyis az anyától), és a spermium ugyan szintén tartalmaz mitokondriumokat – amelyek a mozgásához szükséges energiát adják –, de ezeket megtermékenyítés után már nem örökíti tovább. Így aztán, bár az Y kromoszómától eltérően az anya minden gyerekének átadja mtDNS-ét, az a fiúkkal már nem tud továbböröklődni, azaz genetikai zsákutcába kerül.
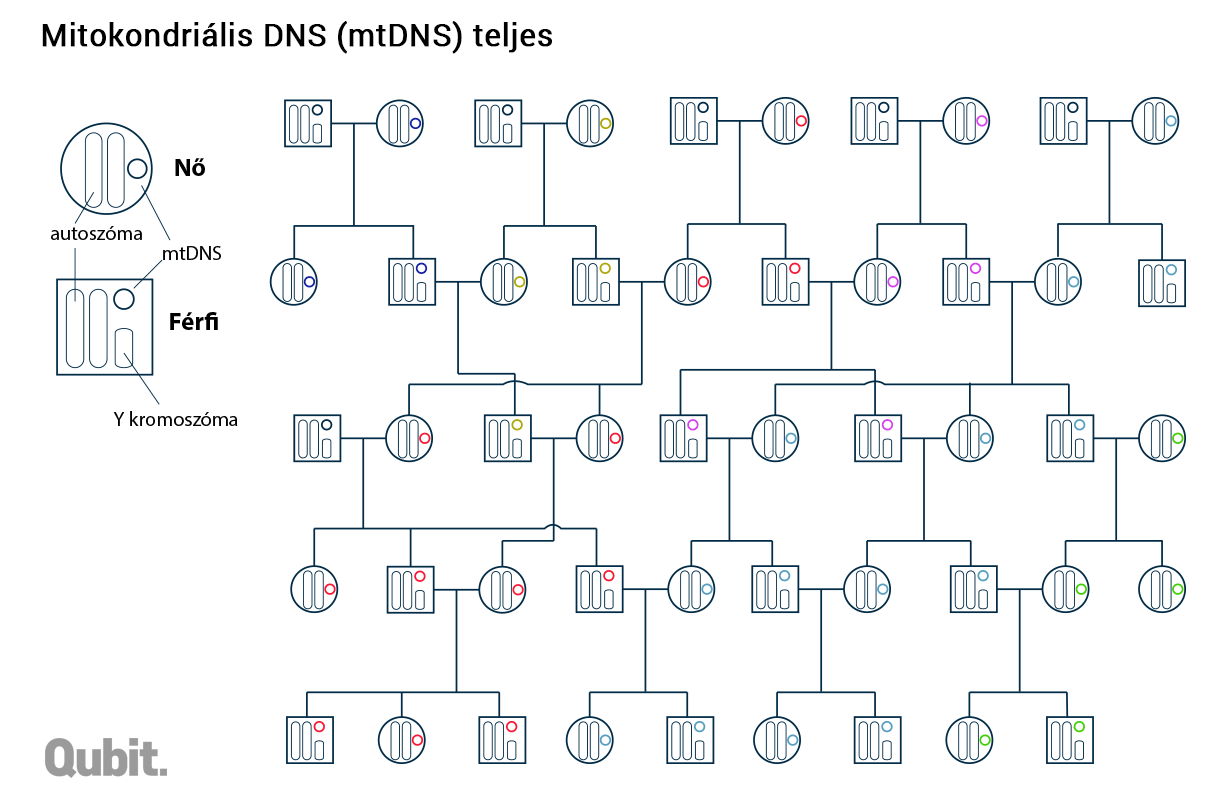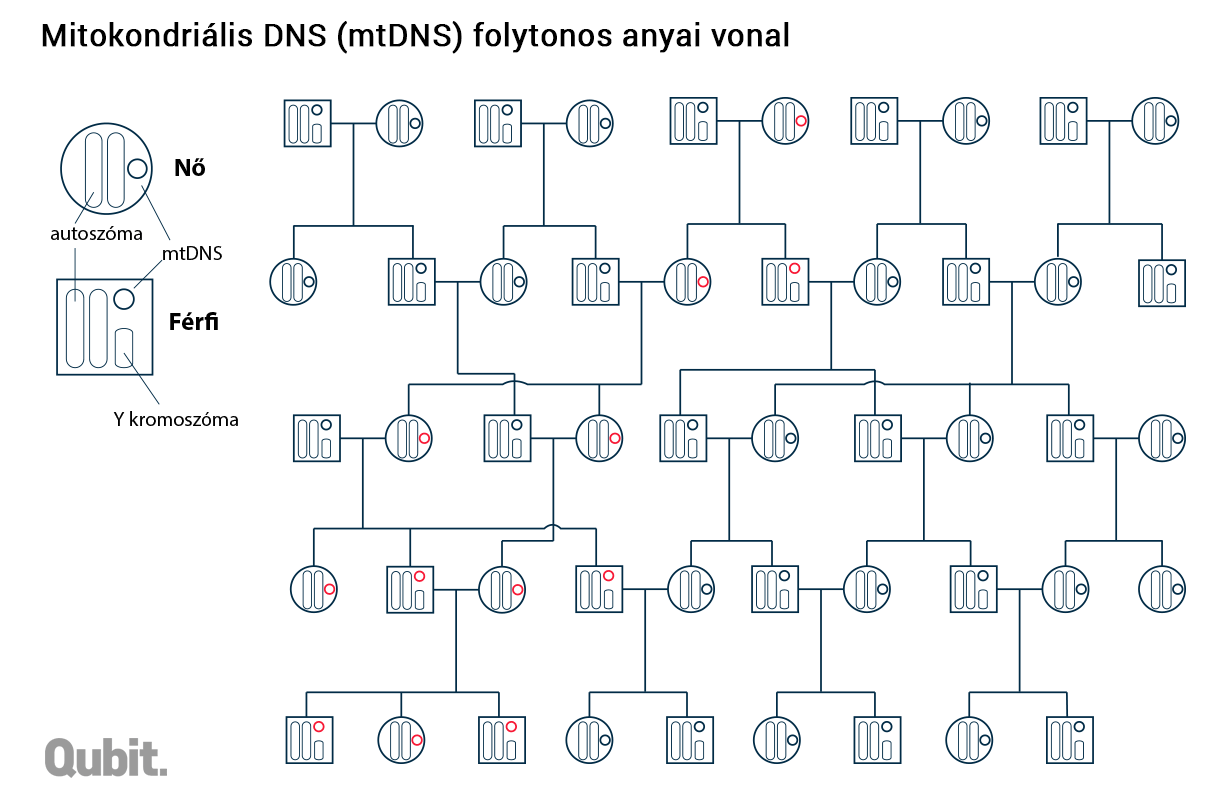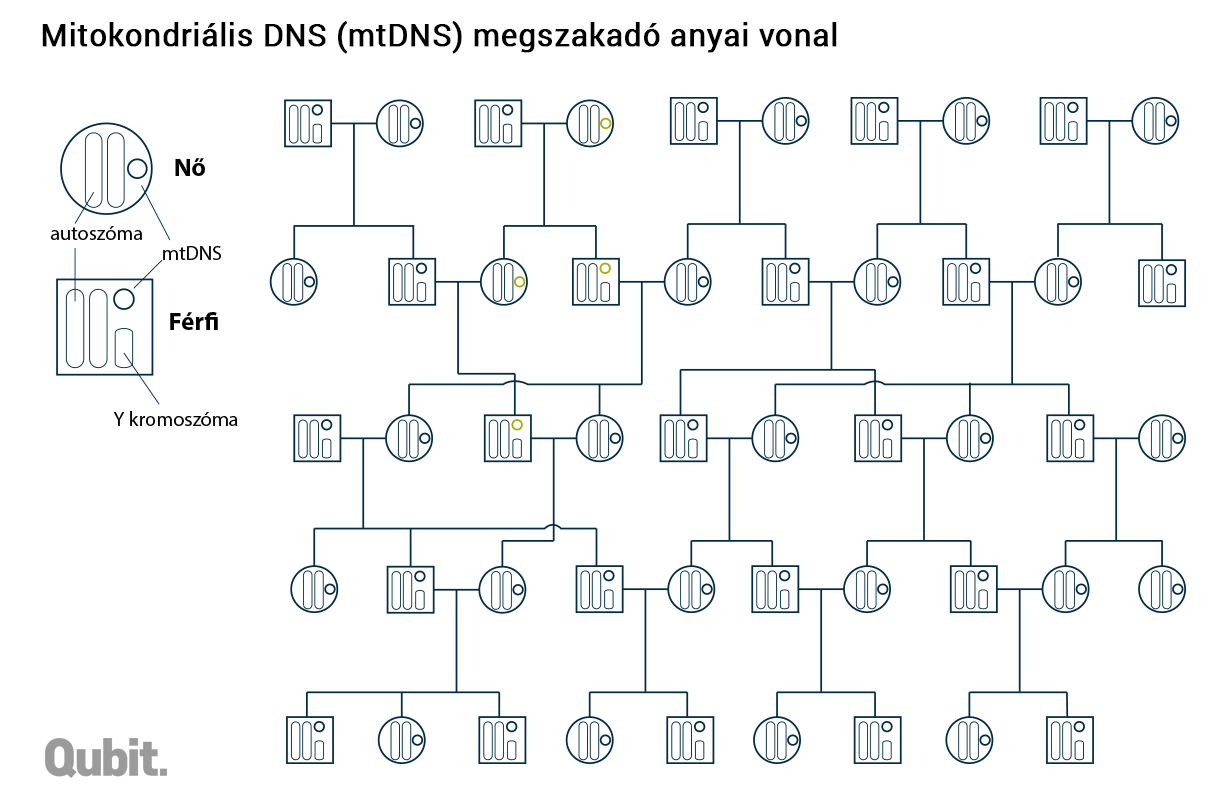
DNS-be rejtett őstörténet
Különleges öröklődésük következtében az mtDNS és az Y kromoszóma alkalmas a matrilineáris, illetve patrilineáris vonalak felderítésére. Ez erősen limitált információ, hiszen segítségükkel a 250 évvel ezelőtt élt ezernyi ősünk közül mindössze kettőről kaphatunk információt. Az Y kromoszóma egy diploid genom kicsit kevesebb mint 1 százalékát hordozza, a rövidke mtDNS pedig a sejtmagban levő genetikai információnak mindössze 0,00027 százalékát. Viszont mivel a rekombináció nem variálja folyamatosan a szekvenciájukat, a patri- illetve matrilineáris vonalon ez a két DNS-molekula kvázi változatlan.
A genetikai változatosság egyetlen forrását a DNS másolása során fellépő hibák jelentik. Mint a kéziratok sokszorosítását végző középkori kódexmásolók, úgy a DNS replikációját végző fehérjék is hibáznak – az mtDNS egyébként alig, legfeljebb ha egymillió „karakterenként” egyszer. Amennyiben ez a tévedés (mutáció) nem nagyon káros, akkor a természetes szelekció nem szórja ki, fennmaradhat és öröklődhet.
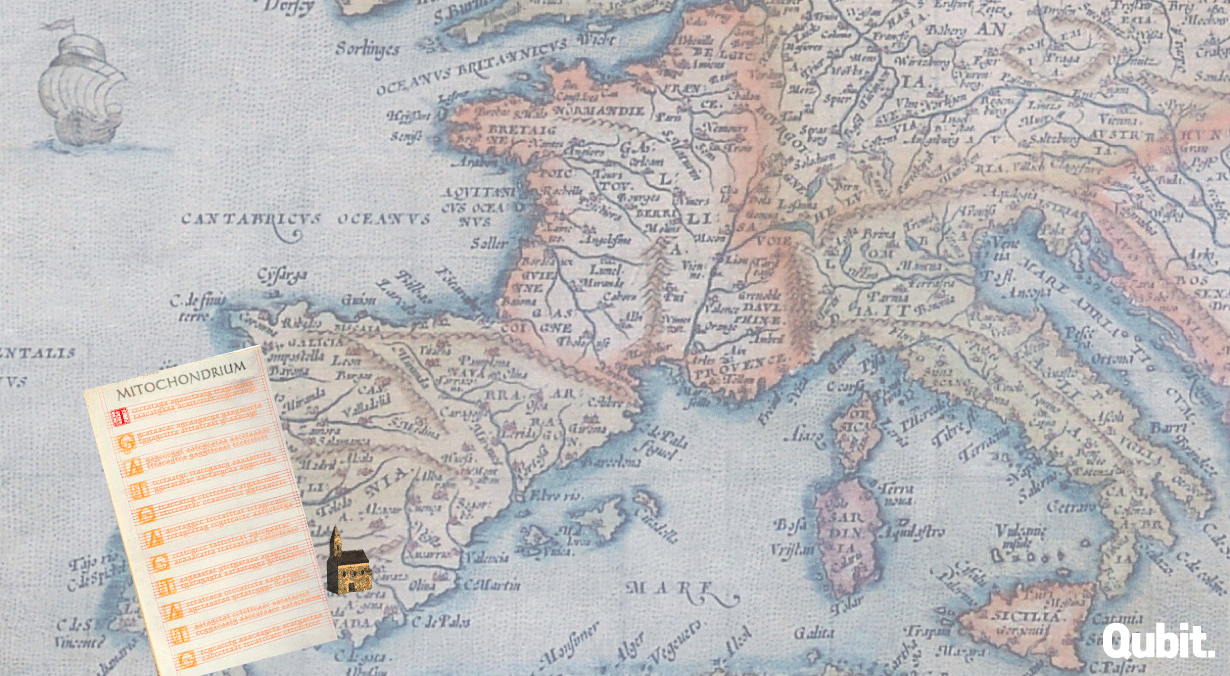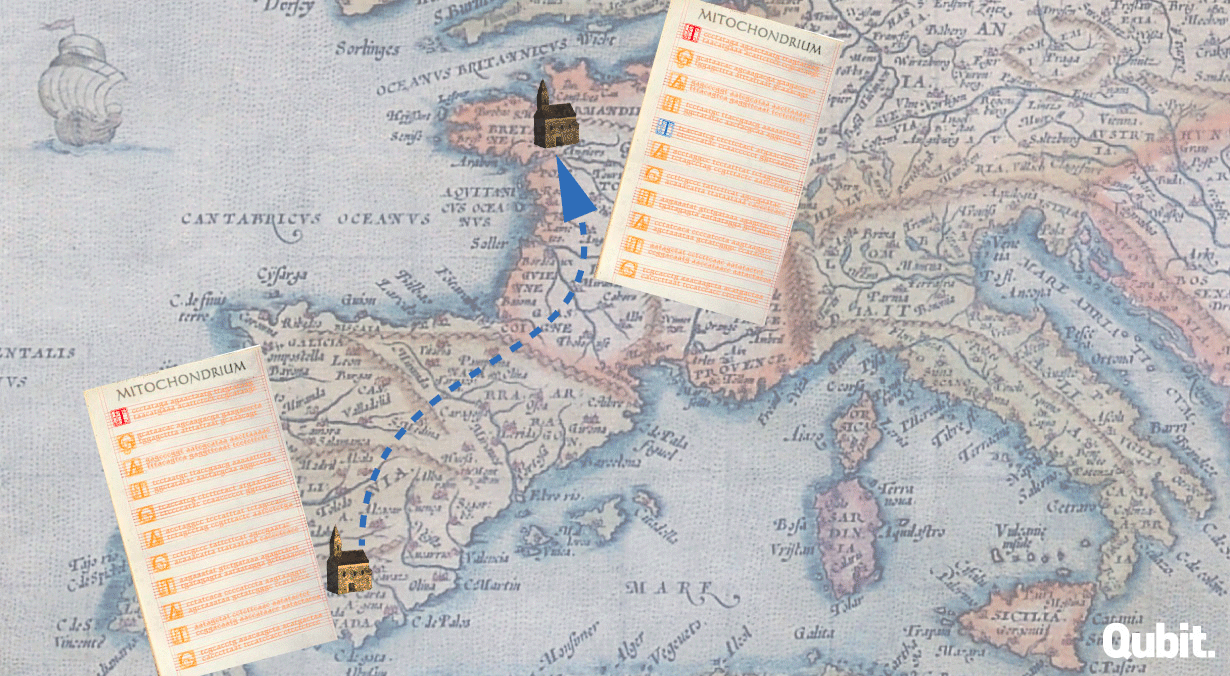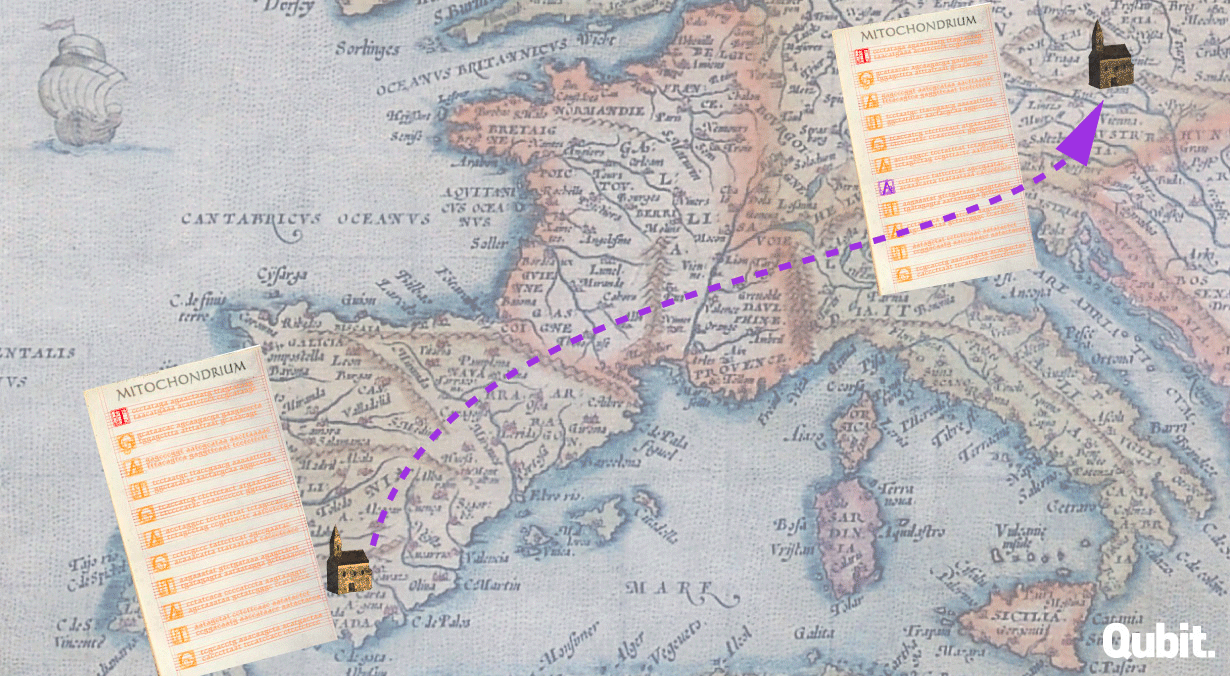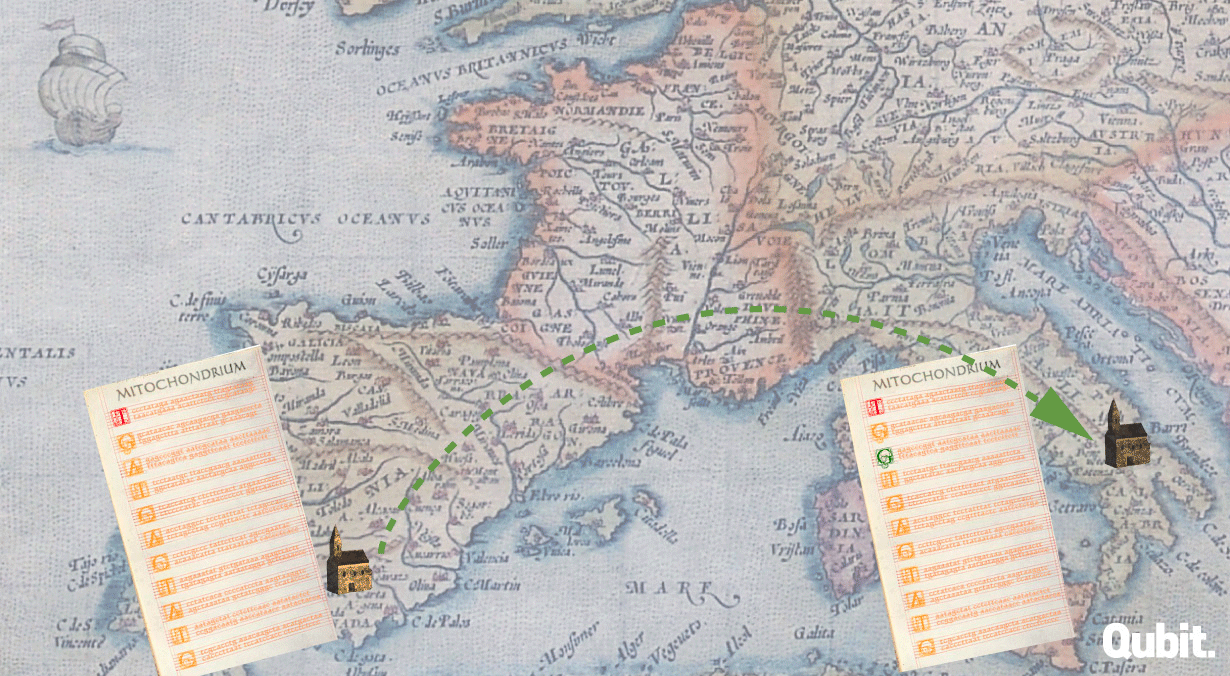
Ibn Rusdnál és a kéziratmásolós hasonlatnál maradva, képzeljük el, hogy valamikor a 15. században egy spanyol kolostorban az Európában később csak Averroesként ismert andalúz filozófus eredeti szövegét másoló írnok hibát vét, de nem teszi érthetetlenné a szöveget, így ezt később sem veszi senki észre. A hibás másolatról készült (immár szintén hibás) másolatok elkerülnek olasz, francia és német kolostorokba is, ahol szorgalmas (de nem tévedhetetlen) szerzetesek másolják a szöveget, eltérő helyeken más és más hibákat vétve. Így ugyan a spanyol kolostorban vétett hiba mindegyikben jelen lesz, ám az újabb hibák már különböznek. E folyamat generációkon át ismétlődik. Az utókor képviselőjeként mi már csak azzal szembesülünk, hogy a világban nagyon sok hasonló tartalmú ibn Rusd-nak tulajdonított mű létezik, de jellegzetes különbségek vannak köztük. Ha viszont felfejtjük és megértjük, hogy ezek a különbségek milyen folyamatnak köszönhetőek, akkor a ma is elérhető szövegek összehasonlításával többé-kevésbé rekonstruálni tudjuk, milyen volt Averroes eredeti szövege, még ha a legősibb verziók el is vesztek. Nem működik ez másképp akkor sem, ha DNS-szekvenciákat tanulmányozunk.
Ráadásul, mivel a mutációk viszonylag egyenletes sebességgel jelennek meg a genomban, a világ különböző részein élő emberek mtDNS-ét (vagy Y kromoszómáját) összevetve a különbségek alapján többé-kevésbé megbecsülhető, hogy mikor (és hol) váltak szét a matri- és patrilineáris vonalak. És az is, hogy hol élt az a személy, akitől minden ma élő ember mtDNS-e, illetve minden ma élő férfi Y kromoszómája származik. Ők azok, akiket a populáris irodalomban „mitokondriális Évaként”, illetve „Y kromoszómás Ádámként” emlegetnek, és akik a legvalószínűbben valamikor 100-170 ezer évvel ezelőtt éltek valahol Kelet Afrikában – de nem feltétlenül egy időben.5
(Ugyan az Y kromoszóma tanulmányozása is rendkívül informatív, a következőkben praktikus okokból mégis az mtDNS-re koncentrálunk. Egyrészt, mert nemtől függetlenül mindenkiben megtalálható, másrészt az ősi emberi maradványokból kinyert archaikus DNS tanulmányozása során nagyon hasznosnak bizonyult, hogy mivel egyetlen sejtben akár ezer mitokondrium is lehet – amelyek mindegyike tartalmazza a minimum egy mtDNS-molekulát –, az mtDNS természetes módon is felsokszorozott formában van jelen a sejtekben. Így viszont még akkor is elég jól rekonstruálható a nukleotid sorrendje, amikor a maradványokra ható természetes folyamatok már akár a teljes sejtmagi DNS mindkét példányát a felismerhetetlenségig összetörték.)
És akkor jönnek a haplotípusok
A „mitokondriális Éva” visszakövetése azonban csak egy a nagyszámú mtDNS szekvenciák összevetésével kapható látványos eredmények közül. Legalább ennyire izgalmas, hogy az Afrikából kiáramló, anatómiailag modern emberek vándorlási útvonalát és főbb állomásait is tudjuk rekonstruálni a módszerrel. Állandó hasonlatunkkal élve, az Averroes-szövegváltozatok összevetésével rekonstruálhatjuk, hogy egy német kolostorba a spanyol másolat egy francia kitérő után jutott el, (tartalmazza ugyanis a francia írnok hibáit), míg az olasz műhelybe egyenesen Hispániából érkezett a szöveg. A kéziratváltozatok hibái jellegzetes mintázatot alkotnak. Nincs másképp ez az mtDNS esetében sem: a különböző mutáció-kombinációk mintázata hozza létre azt, amit a szakzsargon haplotípusnak nevez. A ma megfigyelhető haplotípusok egy nagy leszármazási fába rendezhetők, amely alapján megsaccolható, hogy az egyes mutációk mikor és hol jelentek meg, valamint az is, hogy milyen ütemben terjedtek.
Bő évtizede használatosak azok a genetikai kitek (tesztkészletek), amelyek az mtDNS haplotípusát meghatározva jó pénzért megmondják, hova valósi matrilineáris vonalunk őse. (Számos, teljesen homályban maradt felmenőnkkel szemben erről a személyről legalább tudjuk, hogy valóban genetikai ősünk, így persze hajlamosak vagyunk azonosulni is vele, pedig az ezen DNS-darab által kódolt – a mitokondrium működéséhez fontos – 37 gén aligha határoz meg bármiféle komplexebb identitást.)
A haplotípusok kialalakulását tanulmányozva hangsúlyozni kell, hogy önmagukban nem rendelhetők diszkréten körülhatárolható földrajzi egységekhez. Meghatározhatjuk ugyan azt a területet, ahol az adott haplotípus a leggyakoribb (és ahol valószínűleg kialakult, bár ez sem mindig egyértelmű), de ugyanilyen fontos, hogy miként változik innen távolodva a gyakorisága. Ami persze azt is jelenti, hogy igazából nem létezik olyan emberi populáció, ahol egyetlen mtDNS kizárólagos lenne. Bonyolítja a megértést, hogy ugyanakkor minden ma élő populációt konkrét haplotípus-eloszlások jellemeznek.
Neandervölgyi halhatatlanság
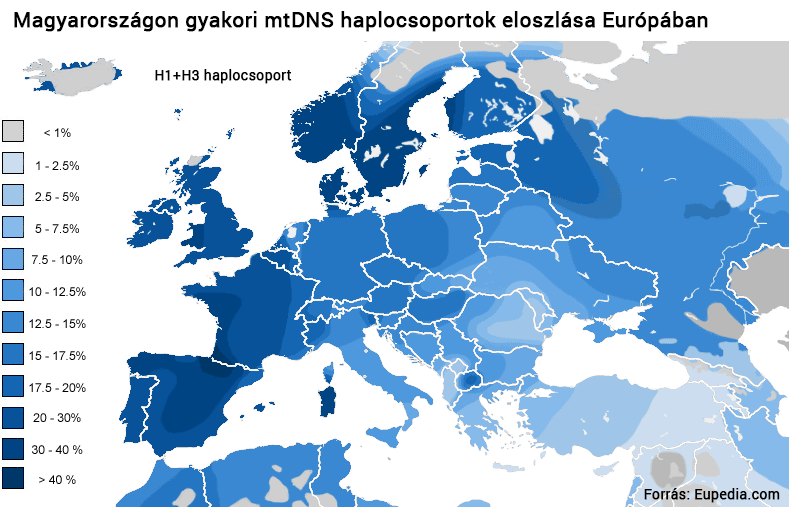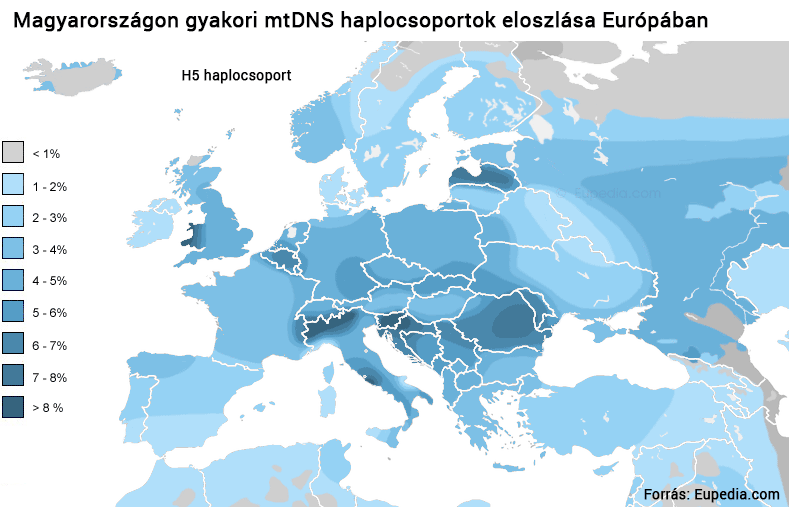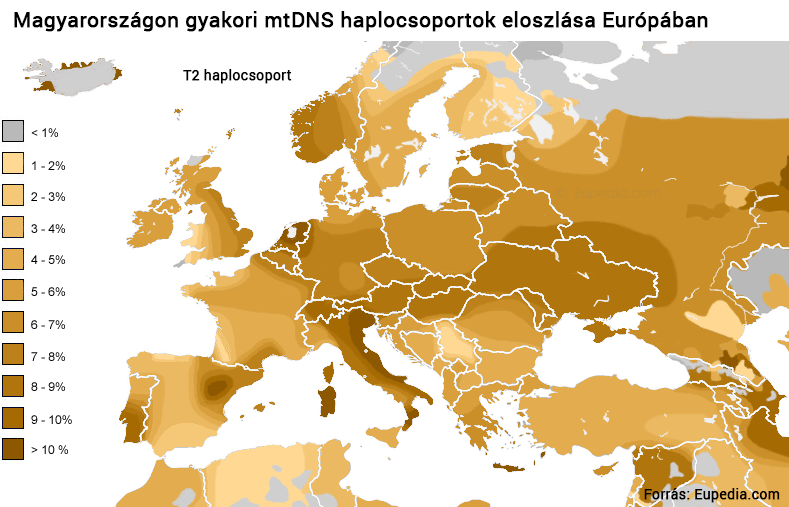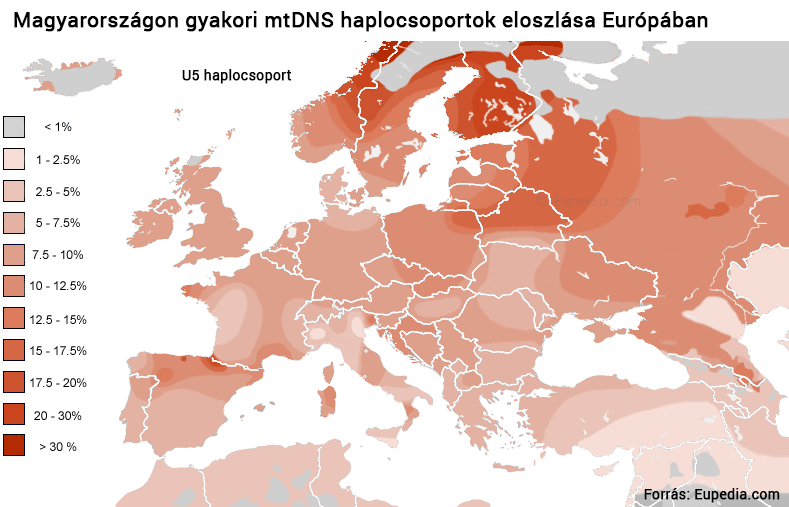
A mai Magyarországon négy haplocsoport (H, J, T2, U5) adja a teljes mitokondriális diverzitás közel kétharmadát.6 Nincs ez másképp a szomszédos országokban sem, ahol ugyanezek a leggyakoribbak, bár a gyakoriságokban természetesen megfigyelhető pár százalékos ingadozás. Az Európában leggyakoribb H haplocsoport (a hazai teljes mtDNS közel 40 százaléka) jelenkori eloszlása alapján sok kutató valószínűsíti, hogy az ősi verzió valamikor 25-30 ezer éve jött létre a Mediterráneum keleti részében, ahol még ma is meghatározó. Hordozói innen érkeztek az európai kontinensre a legutóbbi jégkorszak előtt. A jégtakaró terjeszkedése miatt nem sokkal később azonban már csak néhány lakható régió maradt a földrészen. A vadászó-gyűjtögető életmódot folytató embercsoportok ezekbe az úgynevezett refúgiumokba húzódtak vissza, ahonnan az enyhülést hozó klímafordulatot követve benépesítették egész Európát. A jégkorszaki izoláció során az Ibériai-félszigeten a H haplocsoport ősi szekvenciájában számos új mutáció keletkezett, amelyek a kontinens rekolonozációja során terjedtek el. Senki sem vitatja, hogy egy nyugat-kelet irányú gradiens az egykori ibériai refúgiumban létrejövő H al-haplocsoportok esetében (H1 és H3) ma is kimutatható.7
A H haplocsoport térnyerése a másik ősi U5 térvesztésével párhuzamosan történt. (Az ok-okozati kapcsolat a múlt jótékony ködébe vész.) Az U5 már a jégkorszak előtt jelen volt Európában és a jégkorszak idején, sőt, a jégtakaró visszavonulását követően is a kontinens domináns haplocsoportja lehetett egy ideig. Ma azonban mindössze 10 százaléka a teljes európai népesség mtDNS-ének.8 A jégkorszakot követő újranépesülés során Nyugat Ázsiából, illetve a Közel-Keletről migráltak Európába a J és T2 mtDNS haplotípusok, amelyek további 10-10 százalékát adják a kontinens mai mtDNS-összetételének.
Biztos bizonytalan vs. bizonytalan biztos
A szekvenálási költségek radikális csökkenésével bő tíz éve elérhető DNS-tesztekkel operáló cégek a már ismert mtDNS-adatbázisok alapján rajzolták meg a matrilineáris leszármazási fákat, ezekre vetítik rá a kíváncsi megrendelő egyéni adatait – a kuncsaft egyedi szekvenciája és egyéb személyes adatai alapján folyamatosan bővítve és finomítva a módszert. Az iparág mestere lett a színes nyilakkal ellátott térképek készítésének, és az olyan „történeteknek”, amelyek a szóban forgó mtDNS-haplotípusok kialakulásáról regélnek. Ezek a sztorik épp csak azt hallgatják el, hogy a saját őseink megismerésének szempontjából mennyire limitáltak az eszközeink.9
Mondhatjuk-e a J haplocsoportot hordozóról, hogy bevándorló, illetve titulálható-e őshonosnak, aki az U5 haplocsoporthoz tartozik? Aligha. Az mtDNS ugyanis korlátozott információt hordoz. Az ilyen általánosító kijelentések egész biztosan tévesek. A sok száz leszármazási vonalunk közül az mtDNS csak a matrilineárisat hordozza. Vagyis nagy a valószínűsége, hogy pár generációt visszalépve a J haplocsoportot hordozó személy ősei közt lesz U5-ös, mi több, akár az apja is lehetett ilyen. Ám, mivel az apai mtDNS nem adódik tovább, ezt ugye nem is látjuk. Hovatovább az is lehet, hogy a matrilineáris vonalon U5 mtDNS-t öröklő személy minden más közvetlen őse J haplocsoportú volt.
Az mtDNS öröklődésével kapcsolatban nagyon is ül a klasszikus bon mot, miszerint a bizonyíték hiánya, nem a hiány bizonyítéka. Jó példa a lyukra futásra, hogy amikor az első neandervölgyi mtDNS-t töredékében, aztán idővel egészében is szekvenálták, sokan elkönyvelték, hogy nem lehetett, így nem is volt keveredés az anatómiailag modern emberek és a neandervölgyiek között.10 A sejtmagi szekvenciánálás után azonban borult az állítás. Ma már szakmai közhely, hogy az európaiak 2-4 százalékban neandervölgyi DNS-t hordoznak genetikai állományukban.11
Az mtDNS-szekvencia alapján megállapított rokonsággal más gond is akad, annak ellenére, hogy a betűről betűre megegyező teljes mtDNS szekvencia a legtutibb ismertetője (kellene legyen) a közeli rokonságnak. Csakhogy a „közeliség” értelmezéshez nem árt tudni, hogy az új mtDNS-mutációk rögzüléséhez száz, vagy még több generáció is kell: valójában még a tökéletes egyezés sem garantálja, hogy az eképpen meglelt rokon közvetlen testvérünk, vagy egy kétezer éve élt anyai felmenőnk.12
Avarok és más válogatott vándornépek
Amikor már nem létező népcsoportok és a ma élők rokonsági viszonyait mtDNS alapon vizsgálják, további problémát generál, hogy gyakran alig-alig van információnk arról, hogy a feltételezett véreink populáció szinten milyen mtDNS-összetétellel rendelkeztek. Ennek hiányában viszont szintén könnyű eltévedni. Karikírozva: a magyar lakosságnak mindössze három ezreléke hordozza az Afrikában domináns L mtDNS haplotípust, de ez így is közel 30 ezer embert jelent. Vagyis, ha nem lenne semmi ismeretünk a magyarországi mtDNS-eloszlásról, és az információért megszekvenált – véletlenszerűen kiválasztva – személyek mindegyikének mtDNS-e L-haplotípusúnak bizonyulna, annak ellenére fals eredményre jutnánk, hogy betartottuk az elvárható tudományos protokolt.
Nem könnyíti meg az sem az mtDNS-adatok elemzését, hogy a történelem tele van olyan példákkal, amikor a nőket lényegében politikai célok érdekében áruba bocsátották, vagy egyszerűen elrabolták, illetve olyan vándorlási eseményekben sem szűkölködünk, ahol a vándorló, később megtelepülő populáció döntő mértékben férfiakból állt. (Saját történeti hagyományunkból gondolhatunk itt az alán hercegnőket elrabló Hunor és Magor mondájára, vagy a kazár feleséggel rendelkező Levedi esetére.) Így korábban az adott népességre nem jellemző mtDNS-szekvenciák kerülnek be a népesség genetikai állományába, ahol a későbbiekben nem csak rögzülhetnek, de el is terjedhetnek.
Mindezek jól mutatják, hogy adott népesség genetikai állományát gyakran véletlenszerű események, vándorlások formálják a történelem során. Hol jelenik meg egy mutáció, kinek hány gyereke lesz, milyen neműek ezek az utódok, milyen géneket örökölnek, mennyire lesznek sikeresek maguk is, ha és amikor szaporodó képes korba élnek. Mindez azt jelenti, hogy eleve nem könnyű rekonstruálni eseményeket (nagyobb adathalmazok azért valamelyest segítenek), mert egy adott történelmi lánc tulajdonképpen sokfajta genetikai mintázatot létrehozhat a későbbiekben, illetve vice versa, közel azonos ma észlelhető mintázatokhoz sok különböző úton juthatunk el. Ezek közül törvényszerűen csak egy lehet helyes, de sokszor nem tudjuk biztosan megmondani, hogy melyik, jobb esetben is csak a legvalószínűbbeket tudjuk azonosítani.
Hun a magyar?
A honfoglalók genetikai állománya több évtizede a közbeszéd slágertémája, mondhatni tízmillió archeogenetikus országa is vagyunk.
Szegedi és budapesti kutatóknak köszönhetően lassanként egyre több kirakódarabka került a helyére a honfoglalók, illetve a Kárpát-medence őshonos (valószínűleg elszlávosodott) avar populációinak genetikai összetételéről – a honfoglalás legvalószínűbb forgatókönyve pedig a korábbihoz képest részletgazdagabb lett, de meg is változott.
Több tucatnyi avar és közel száz honfoglaló mtDNS-szekvencia alapján az látszik, hogy a mai magyar népesség genetikai összetételében jobban hasonlít a népvándorlás előtt e tájékon élő avarokhoz, mint az e szempontból is diverz honfoglalókhoz.13 E sokszínűség nem meglepő: az őstörténet alapján nyilvánvaló, hogy hosszú vándorlás, számos néppel való interakció, illetve az idegen törzsek integrálása során jött létre az a genetikai konglomerátum, amit az utókor „honfoglaló magyarnak” aposztrofál. (A kutatásokban regisztrált közel száz mitokondriális haplotípus már egész reprezentatívnak tűnik, ugyanakkor mivel nagy részük mindössze alig pár temetőből származik, nem állíthatjuk teljes bizonyossággal, hogy a minták tükrözik a honfoglalók teljes genetikai skáláját.)
Az értelmezési nehézségek négyzetre emelve jelentkeznek, amikor két, már kihalt népességet hasonlítunk össze – mondjuk a Kárpát-medencét elfoglaló magyarokat és a hunokat.14 Jobb híján hunként fogadunk el olyan mintákat, amelyek ázsiai hsziungnu temetőkből és sírokból kerültek elő, pusztán azért, mert a maradványok mellett az ázsiai hun tárgyi kultúra elemei voltak fellelhetők.15 Nem árt tudni, hogy a sikeres törzsek, népcsoportok ebben az időszakban rendszerint beolvasztották a meghódított/leigázott etnikumokat. Egykori források szerint ez állt a hunokra is, tehát csupán a tárgyi bizonyítékok alapján nehéz megmondani, hogy egy hunként azonosított tárgyakkal eltemetett személy hetedíziglen is hun volt-e, vagy a csatlakozó népek valamelyikéhez tartozott. Ráadásul a „sztyeppei országút” évezredeken genetikai mixerként működött. A kelet-nyugati, illetve nyugat-keleti irányú népmozgások újra és újra összekeverték a környező térségek emberpopulációinak genetikai állományát. A rokonság genetikai bizonyításához, illetve cáfolatához olyan nagy számú DNS-mintára volna szükség, amivel jelenleg nem rendelkezünk – kétségtelenül dűlőre vinné a hun-magyar vitát, ha a honfoglaló törzsek korábbi szálláshelyeinek őslakosságáról is lennének genetikai ismereteink.
A folytatásban felvázoljuk, hogy melyek azok a dolgot, amelyeket mindannyiunk leszármazásáról bizton állíthatunk.
(A szöveg szakmai lektora Szécsényi-Nagy Anna, nyelvi lektora pedig Víg Julianna volt. Külön köszönet munkájukért.)
Lábjegyzetek
- Fontos megjegyezni, hogy egy génnek nem csak egy működőképes és egy működésképtelen allélja létezhet, hanem a teljes népességben mindent összevetve nagyon sok olyan allélt találunk, ami olyan DNS-változatot (polimorfizmust) hordoz, hogy az érdemben nem befolyásolja a gén működését és még több olyan allélt, amely mutációja pedig hatással van a rá, bár eltérő mértékben. A lényeg, hogy egy normális diploid szervezetben mindig csak két allél lesz egymás mellett.
- A rekombináció ugyanis viszonylag hatékonyan és gyorsan egyetlen genomba tud rendezni különböző egyedekben megjelenő előnyös mutációkat. Erre az aszexuálisan (vagyis kvázi önmaguk klónozásával) szaporodó egyedek képtelenek - ott az előnyös mutációknak egyenként kellene ugyanabban a leszármazási vonalban megjelennie. Így aztán a párválasztáshoz tartozó minden szembetűnő jelleg és viselkedés, a pávatolltól, a nyeles szemekig, a szarvasbőgéstől, a fészeképítésig tulajdonképpen mind ennek a szabad szemmel nem is látható folyamatnak a sikerre vivéséről szól.
- Vagyis, amennyiben az SRY nem működik, akkor Y kromoszóma mellett is nővé fejlődik az embrió , illetve, ha valahogy az SRY átkerül egy másik kromoszómára, akkor Y kromoszóma hiányában is kialakulnak a hím jellegek. (Procopiuc 2009; Bashamboo and McElreavey 2015)
- A teljes képhez hozzátartozik, hogy az Y kromoszóma két végén található egy-egy rövid rész, ami képes az X kromoszóma végeivel párba állni - ezeket pszeudoautoszomális régiónak (PAR) nevezzük -, s így a valóságban ugyanúgy működik, mint a többi kromoszóma.
- A pontos időpontja a mitokondriális “Éva” és az Y kromoszómális “Ádám” létezésének nem teljesen lefutott ügy. A bizonytalanságot az adja, hogy különböző becslések alapján kicsit eltérő, hogy mennyire gyorsan keletkeznek és rögzülnek mutációk az mtDNS-ben, vagy az Y kromoszómán, így különböző modellek alapján visszavetítve, eltérő időpontokat kaphatunk. Korábbi eredmények szerint a két ős nagyon eltérő időben élhetett (“Ádám” 100 ezer évvel, “Éva” kétszázezer évvel ezelőtt), de az új, nagyobb adathalmazokon és modernebb módszereken alapuló eredmények közelítettek ezeket a becsléseket.
- Az mtDNS eloszlások adatai az Eupedia adatbázisából származnak.
- A mitokondriális H haplocsoport eredete: (Brotherton et al. 2013); (Hernández et al. 2017). A teljes objektivitás kedvéért itt fontos megjegyezni, hogy a H haplocsoport eredetére vonatkozóan léteznek más magyarázatok is - például a jelenlegi mtDNS variációból visszafele következtető populációgenetikusokkal ellentétben a korabeli mintákat néző archeogenetikusok inkább azt tartják valószínűnek, hogy a H haplocsoport is az első földművesekkel érkezett a Közel-Keletről.
- A mitokondriális U5 haplocsoport eredete: (Posth et al. 2016); (Malyarchuk et al. 2010)
- Az utóbbi években elterjedt sejtmagi DNS-t vizsgáló kitek természetesen már sokkal informatívabbak ilyen szempontból.
- Az első neandervölgyi mtDNS szekvenciák és értelmezésük: (Hodgson and Disotell 2008; Green et al. 2008; Serre et al. 2004)
- A neandervölgyi és mai európai genomok közti hasonlóság 2-4%: (Prüfer et al. 2014; Vernot and Akey 2014; Sankararaman et al. 2014)
- Kb. 100 generációnként egy mutáció az mtDNSben: (Soares et al. 2009)
- Archeogenetikai eredmények honfoglaláskori mintákból: (Csősz et al. 2016; Neparáczki et al. 2017)
- A pár hónapja sok vihart kavart hun-honfoglaló kontinuitás adatait publikáló cikk: (Neparaczki et al. 2018)
- Ismert hsziungnu minták: (Pilipenko et al. 2017; Kim et al. 2010; Keyser-Tracqui, Crubézy, and Ludes 2003)
Hivatkozások
Bashamboo, Anu, and Ken McElreavey. 2015. “Human Sex-Determination and Disorders of Sex-Development (DSD).” Seminars in Cell & Developmental Biology 45:77–83.
Brotherton, Paul, Wolfgang Haak, Jennifer Templeton, Guido Brandt, Julien Soubrier, Christina Jane Adler, Stephen M. Richards, et al. 2013. “Neolithic Mitochondrial Haplogroup H Genomes and the Genetic Origins of Europeans.” Nature Communications 4:1764.
Csősz, Aranka, Anna Szécsényi-Nagy, Veronika Csákyová, Péter Langó, Viktória Bódis, Kitti Köhler, Gyöngyvér Tömöry, Melinda Nagy, and Balázs Gusztáv Mende. 2016. “Maternal Genetic Ancestry and Legacy of 10th Century AD Hungarians.” Scientific Reports 6 (1). https://doi.org/10.1038/srep33446.
Green, Richard E., Anna-Sapfo Malaspinas, Johannes Krause, Adrian W. Briggs, Philip L. F. Johnson, Caroline Uhler, Matthias Meyer, et al. 2008. “A Complete Neandertal Mitochondrial Genome Sequence Determined by High-Throughput Sequencing.” Cell 134 (3):416–26.
Hernández, Candela L., Jean M. Dugoujon, Andrea Novelletto, Juan N. Rodríguez, Pedro Cuesta, and Rosario Calderón. 2017. “The Distribution of Mitochondrial DNA Haplogroup H in Southern Iberia Indicates Ancient Human Genetic Exchanges along the Western Edge of the Mediterranean.” BMC Genetics 18 (1):46.
Hodgson, Jason A., and Todd R. Disotell. 2008. “No Evidence of a Neanderthal Contribution to Modern Human Diversity.” Genome Biology 9 (2):206.
Keyser-Tracqui, Christine, Eric Crubézy, and Bertrand Ludes. 2003. “Nuclear and Mitochondrial DNA Analysis of a 2,000-Year-Old Necropolis in the Egyin Gol Valley of Mongolia.” American Journal of Human Genetics 73 (2):247–60.
Kim, Kijeong, Charles H. Brenner, Victor H. Mair, Kwang-Ho Lee, Jae-Hyun Kim, Eregzen Gelegdorj, Natsag Batbold, et al. 2010. “A Western Eurasian Male Is Found in 2000-Year-Old Elite Xiongnu Cemetery in Northeast Mongolia.” American Journal of Physical Anthropology 142 (3):429–40.
Malyarchuk, Boris, Miroslava Derenko, Tomasz Grzybowski, Maria Perkova, Urszula Rogalla, Tomas Vanecek, and Iosif Tsybovsky. 2010. “The Peopling of Europe from the Mitochondrial Haplogroup U5 Perspective.” PloS One 5 (4):e10285.
Neparáczki, Endre, Klaudia Kocsy, Gábor Endre Tóth, Zoltán Maróti, Tibor Kalmár, Péter Bihari, István Nagy, et al. 2017. “Revising mtDNA Haplotypes of the Ancient Hungarian Conquerors with next Generation Sequencing.” PloS One 12 (4):e0174886.
Neparaczki, Endre, Zoltan Maroti, Tibor Kalmar, Klaudia Kocsy, Kitti Maar, Peter Bihari, Istvan Nagy, et al. 2018. “Mitogenomic Data Indicate Admixture Components of Asian Hun and Srubnaya Origin in the Hungarian Conquerors.” https://doi.org/10.1101/250688.
Pilipenko, Aleksandr S., Stepan V. Cherdantsev, Rostislav O. Trapezov, Anton A. Zhuravlev, Vladimir N. Babenko, Dmitri V. Pozdnyakov, Prokopiy B. Konovalov, and Natalia V. Polosmak. 2017. “Mitochondrial DNA Diversity in a Transbaikalian Xiongnu Population.” Archaeological and Anthropological Sciences. https://doi.org/10.1007/s12520-017-0481-x.
Posth, Cosimo, Gabriel Renaud, Alissa Mittnik, Dorothée G. Drucker, Hélène Rougier, Christophe Cupillard, Frédérique Valentin, et al. 2016. “Pleistocene Mitochondrial Genomes Suggest a Single Major Dispersal of Non-Africans and a Late Glacial Population Turnover in Europe.” Current Biology: CB 26 (4):557–61.
Procopiuc, Camelia. 2009. “Complete Sex Reversal: Sry Positive 46,XX Male by Y to X Translocation.” Acta Endocrinologica 5 (4):525–31.
Prüfer, Kay, Fernando Racimo, Nick Patterson, Flora Jay, Sriram Sankararaman, Susanna Sawyer, Anja Heinze, et al. 2014. “The Complete Genome Sequence of a Neanderthal from the Altai Mountains.” Nature 505 (7481):43–49.
Sankararaman, Sriram, Swapan Mallick, Michael Dannemann, Kay Prüfer, Janet Kelso, Svante Pääbo, Nick Patterson, and David Reich. 2014. “The Genomic Landscape of Neanderthal Ancestry in Present-Day Humans.” Nature 507 (7492):354–57.
Serre, David, André Langaney, Mario Chech, Maria Teschler-Nicola, Maja Paunovic, Philippe Mennecier, Michael Hofreiter, Göran Possnert, and Svante Pääbo. 2004. “No Evidence of Neandertal mtDNA Contribution to Early Modern Humans.” PLoS Biology 2 (3):E57.
Soares, Pedro, Luca Ermini, Noel Thomson, Maru Mormina, Teresa Rito, Arne Röhl, Antonio Salas, Stephen Oppenheimer, Vincent Macaulay, and Martin B. Richards. 2009. “Correcting for Purifying Selection: An Improved Human Mitochondrial Molecular Clock.” American Journal of Human Genetics 84 (6):740–59.
Vernot, Benjamin, and Joshua M. Akey. 2014. “Resurrecting Surviving Neandertal Lineages from Modern Human Genomes.” Science 343 (6174):1017–21.
