Tervezz vadonatúj dizájnfehérjét a saját neveddel!

Tetszőleges aminosav-szekvenciákból képes fehérjemolekulákat generálni egy új fehérjetervező algoritmus, ami kifejlesztőinek reményei szerint lehetővé teszi majd olyan funkcionális fehérjék előállítását, amelyek a kutatók által kívánt jellemzőkkel rendelkeznek. Bónuszként, ha szerencsések vagyunk, most először saját nevünket is beleírhatjuk egy általunk tervezett fehérjékbe.
A Sidney Lisanza, a seattle-i Washingtoni Egyetem Fehérjetervező intézetének biokémikusa és kollégái által május elején egy bioRxiv-ra feltöltött preprintben ismertetett ProteinGenerator egyszerre képes előállítani új fehérjeszekvenciákat és hozzájuk tartozó háromdimenziós struktúrákat, amik megadott fizikai és biokémiai tulajdonságokkal rendelkeznek. Mivel az élethez nélkülözhetetlen molekulák funkcióját háromdimenziós szerkezetük határozza meg, az új, a természetben nem előforduló fehérjeszerkezetek tervezése egyes kutatók szerint újfajta terápiákhoz, valamint a biológiai folyamatok jobb megismeréséhez vezethet.

Tavaly szintén a Washingtoni Egyetem kutatói mutatták be a ProteinMPNN algoritmust. Amint korábbi cikkünkből kiderül, a fejlesztés az addigi módszereknél akár 200-szor gyorsabban és nagyobb eséllyel generált működő fehérjéket. Arne Elofsson svéd bioinformatikus akkor a Nature-nek azt mondta, hogy „a ProteinMPNN-hez hasonló mélytanulási módszerek átírták a játékszabályokat. Megrajzolod a fehérjédet, megnyomsz egy gombot, és kapsz valamit, ami tízből egyszer működik.”
A ProteinMPNN vagy az idén május elején bemutatott ProteinSGM fehérjetervező algoritmus nem volt elérhető a nagyközönség számára. Más a helyzet a ProteinGeneratorral, amit egy interaktív webes felületen bárki kipróbálhat, és ehhez nem is kell molekuláris biológusnak lenni. Elég, ha kiválasztjuk az opciók közül az egyéni szekvenciát takaró „custom sequence” lehetőséget, majd az oldal aljára görgetünk, és rákattintunk a négy közül a legalsó példára.
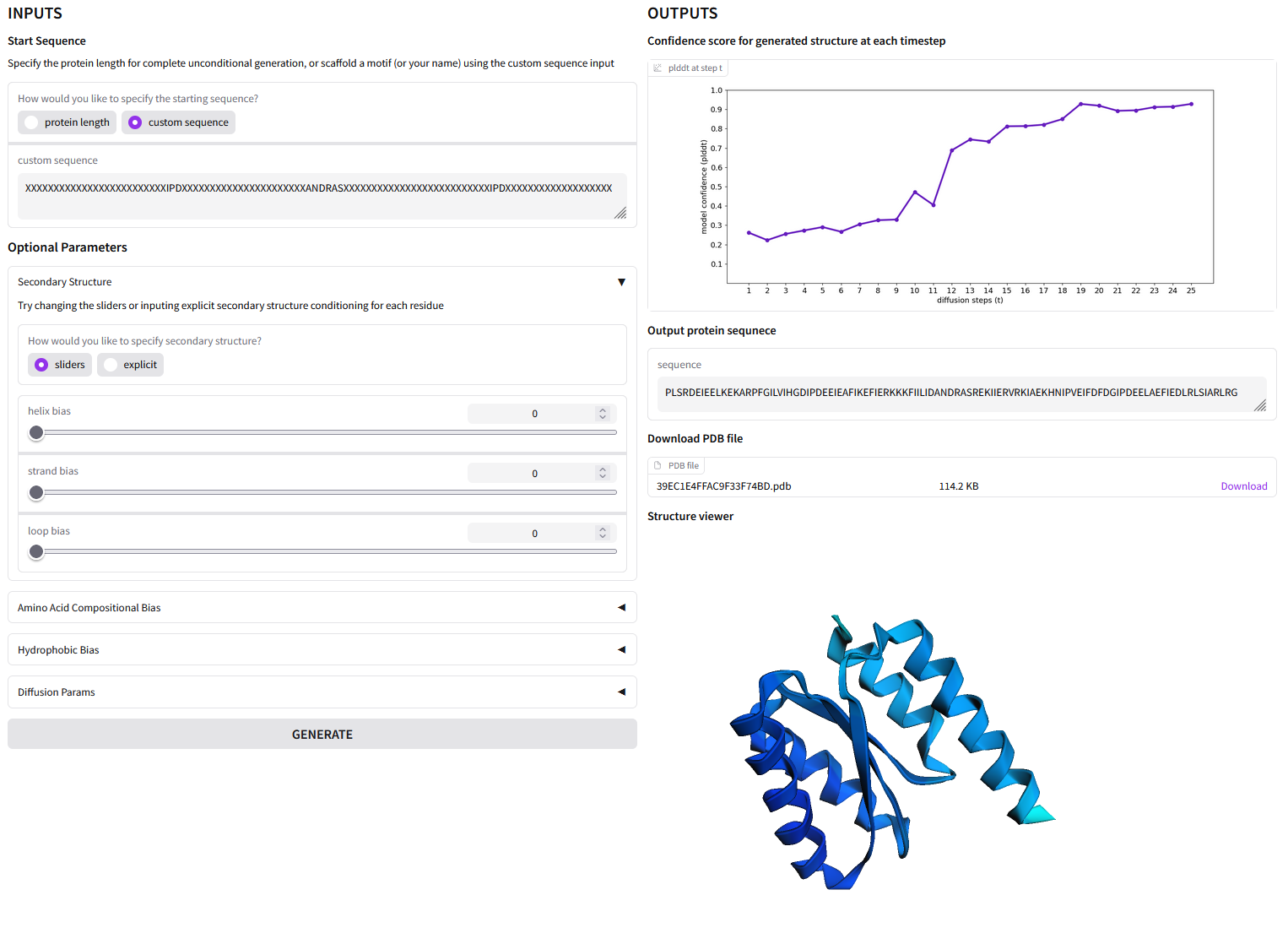
Ezután az időközben megjelenő „custom sequence” dobozban lévő karakterek között kicseréljük a peptidszekvenciára utaló „PEPSEQ” kifejezést a nevünkre (esetünkben: ANDRAS). Természetesen csak a 20 genetikailag kódolt aminosav egybetűs szimbólumát használhatjuk (A, C, D, E, F, G, H, I, K, L, M, N, P, Q, R, S, T, V, W, Y), ami messze van a magyar ábécé 40 betűjétől, de ha ezt a korlátot leküzdöttük, meg is nyomhatjuk a „Generate” gombot.
Innentől másodpercekre vagyunk attól, hogy megszülessen az általunk tervezett fehérje, amin az algoritmus egy ideig finomít még. Az elénk táruló térbeli szerkezet egy háromdimenziós szalagmodell lesz, ahol a fehérjegerinc alfa-hélix régióit feltekert, a beta-láncokat pedig az őket összekötő szalagok szemléltetik. Ezt a térszerkezetet az aminosavak sorrendje kódolja, amit feljebb szintén megkapunk, benne saját nevünkkel, ami így ki tud fejeződni a háromdimenziós struktúrában. A térbeli szerkezetet a PDB fehérjeadatbázis formátumában le is tudjuk tölteni, és a Nanome alkalmazás, valamint egy VR-szemüveg segítségével akár háromdimenzióban is meg tudjuk nézni.

Az új fehérjék tervezéséhez használt DDPM (denoising diffusion probabilistic models) algoritmusok az OpenAI DALL-E-2 képgenerátorához hasonlóan működnek: egy diffúziós modellt használnak, csak nem káprázatos illusztrációkat gyártanak, hanem fehérjéket. Erre, mint a ProteinSGM példáján Ava Amini és Kevin Yang a Nature Computational Science-ben nemrég elmagyarázták, a modell úgy képes, hogy az úgynevezett fehérje-főlánc képszerű reprezentációját a benne található atomok térbeli koordinátái és kémiai kötéseiknek szögei alapján generálja le. A fehérje-főláncok nagyrészt ismétlődő elemekből állnak, és nem tartalmazzák a fehérjék változatosságáért felelős aminosav-oldalláncokat.
Lisanza és kollégái szerint a DDPM algoritmusok rendkívül ígéretes megoldást jelentenek a fehérje-főláncok létrehozására, de képességeikben annyiban korlátozottak, hogy képtelenek specifikus szekvencia- vagy funkcionális jellegekkel rendelkező fehérjék előállítását vezérelni. A ProteinGenerator esetében a kutatók ezt úgy oldották meg, hogy a programot egy fehérjekutató algoritmusra, a RoseTTAFold-ra építették. Ez a DeepMind 2020-ban áttörést hozó AlphaFold2-jéhez hasonlóan szekvenciájukból képes meghatározni, hogyan tekerednek majd fel a fehérjék, és milyen lesz a térbeli, élettani funkciókat ellátó szerkezetük.
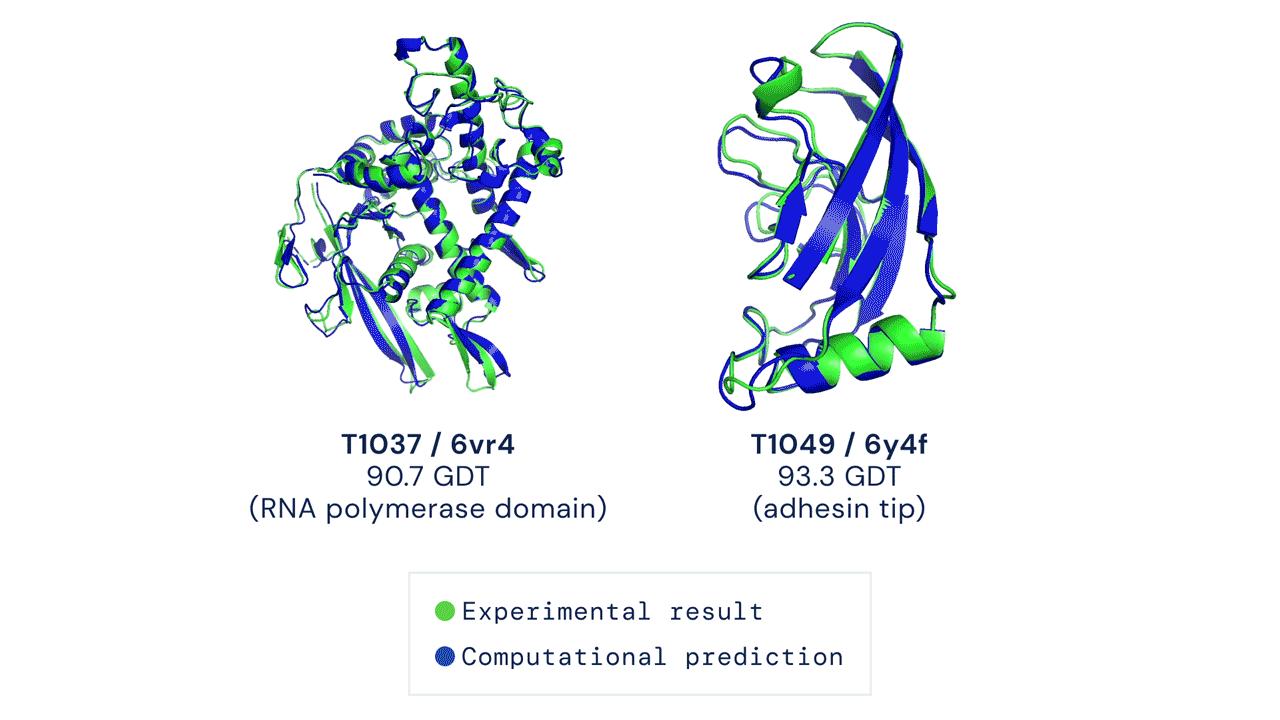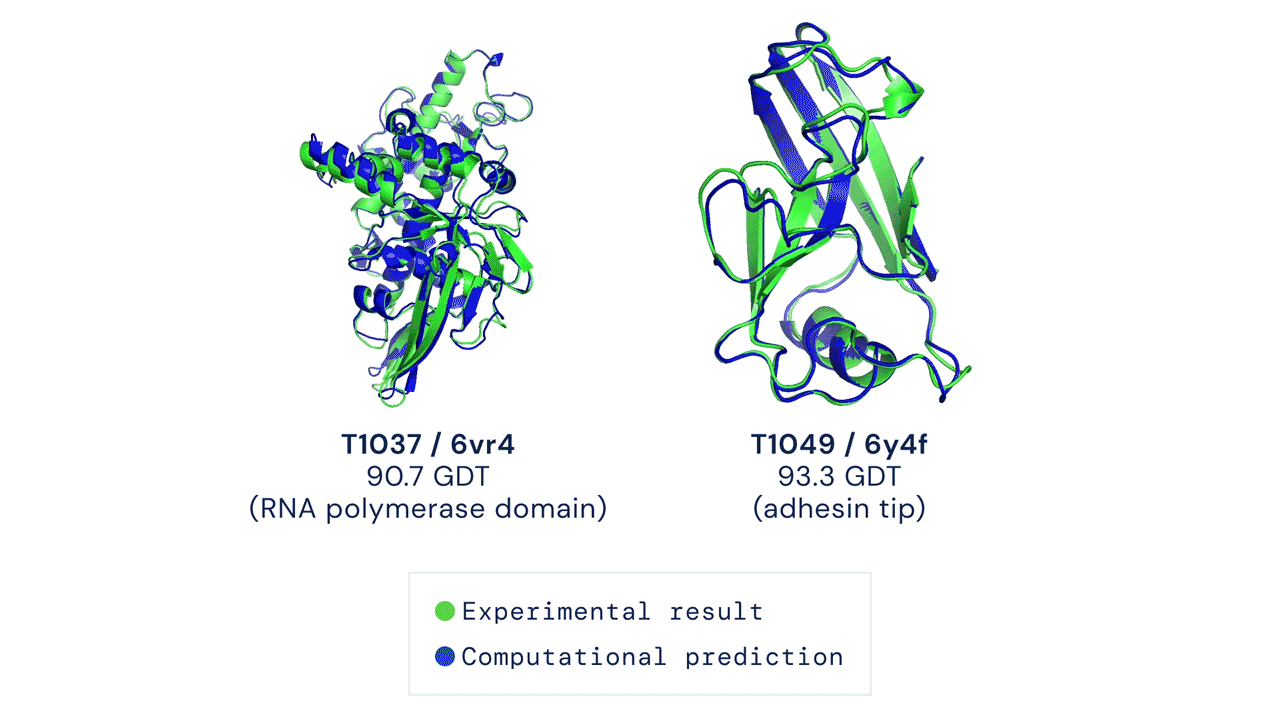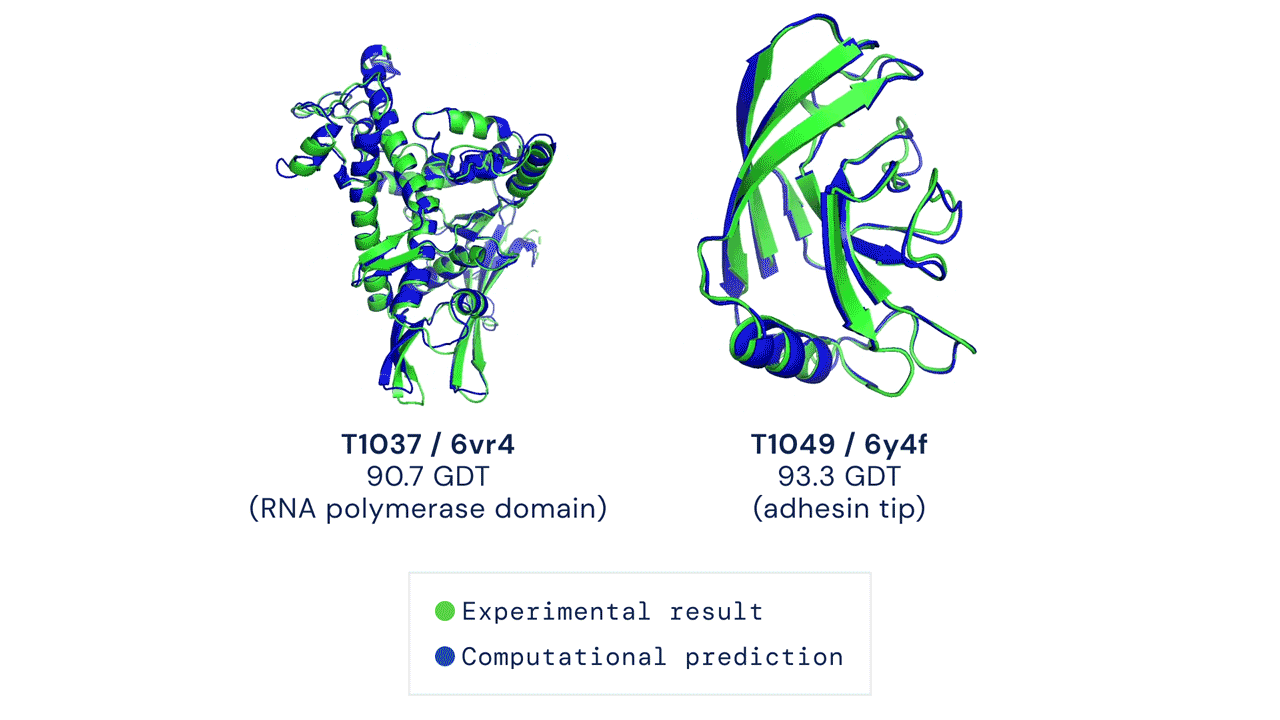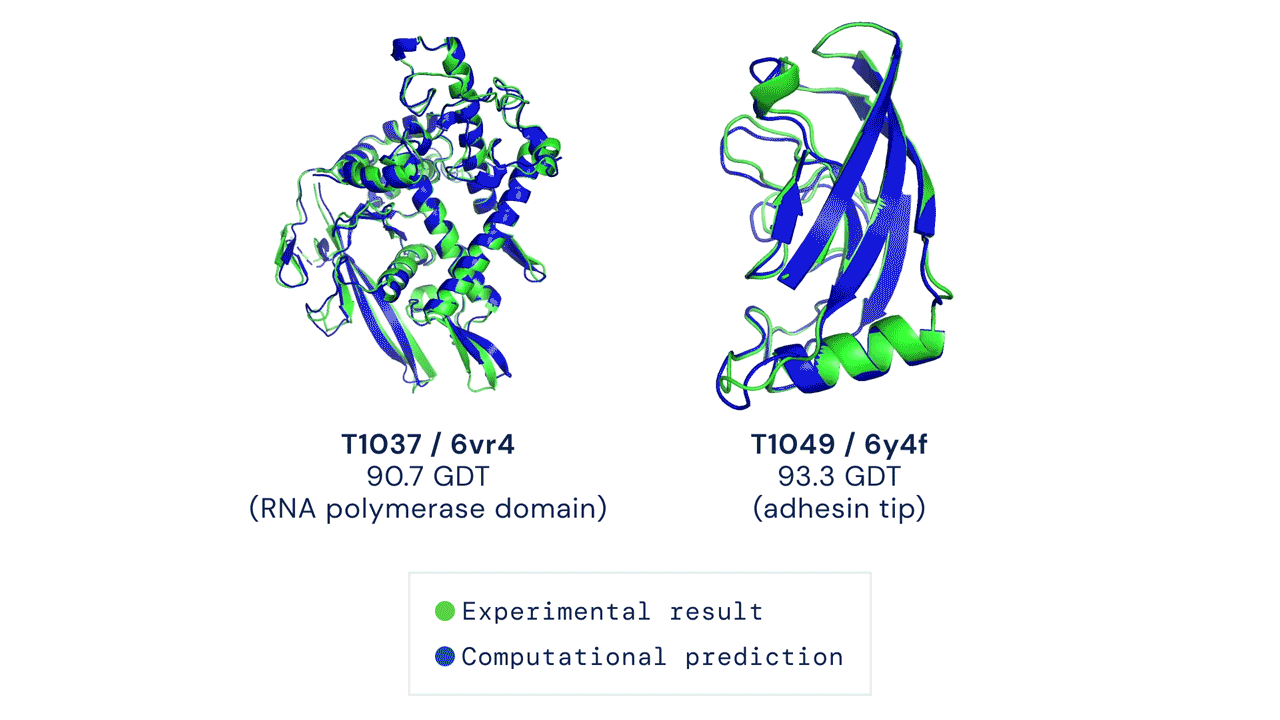
A ProteinGenerator random aminosavszekvenciákból indul ki, és generál le iteratív módon szekvencia-struktúra párosokat úgy, hogy a folyamatot a kívánt szekvencia és strukturális tulajdonságok vezérlik. A kutatók kísérletileg is igazolták az algoritmus képességeit, ami megmutatta, hogy a tervezett és E. coli baktériumokban kifejeztetett, 70-80 aminosav-maradékból álló fehérjék a kívánt térszerkezetet vették fel, és 95 fokos hőmérsékletig stabilnak bizonyultak.
A következő lépés Lisanza szerint létező fehérjecsaládok változatosabbá tétele lehet, amit el is kezdtek a zöld fluoreszcens fehérje (GFP) aktív helyének módosításával. Ennek eredményét hamarosan a laborban fogják tesztelni. Mivel a szintetikus biológia korában élünk, valószínűleg nem kell már sokáig várni, amíg a ProteinGeneratorral és utódaival terveztetett fehérjéinket biohacker terekben kifejeztethetjük különböző élőlényekkel.
Kapcsolódó cikkek a Qubiten:
Elképesztően fejlett fehérjekutató algoritmusok versengenek egymással, hogy feltérképezzék a legalapvetőbb biológiai folyamatokat
A fehérjék szekvenciájából 3D szerkezeteket előállító AlphaFold2 mesterséges intelligencia 2020-ban megoldotta a molekuláris biológia egyik legnagyobb problémáját. Az algoritmusok legújabb versenyén az is körvonalazódott, hogy merre halad tovább a terület.

Soha nem látott fehérjéket álmodik meg a ProteinMPNN mesterséges intelligencia
Az új mélytanulási algoritmus forradalmasíthatja a terápiákhoz és nanotechnológiás szerkezetekhez szükséges fehérjék tervezését. A ProteinMPNN sokkal gyorsabb, mint a korábbi eszközök, és az általa tervezett molekulák többnyire a valóságban is működnek.
A szintetikus biológia korában élünk, de a mesterséges élet létrehozása még mindig csak álom
Az elmúlt húsz év áttöréseinek köszönhetően mára lehetővé vált az élőlények, köztük az ember genomjának szerkesztése, de minden igyekezet ellenére még nagyon messze vagyunk attól, hogy teljesen szintetikus sejtet állítsunk elő.
Kapcsolódó cikkek

![A nagy képrobotcsata: [imagine] Imagine!](https://assets.4cdn.hu/kraken/7tHbHIW3ZSrGNUDAs.png)