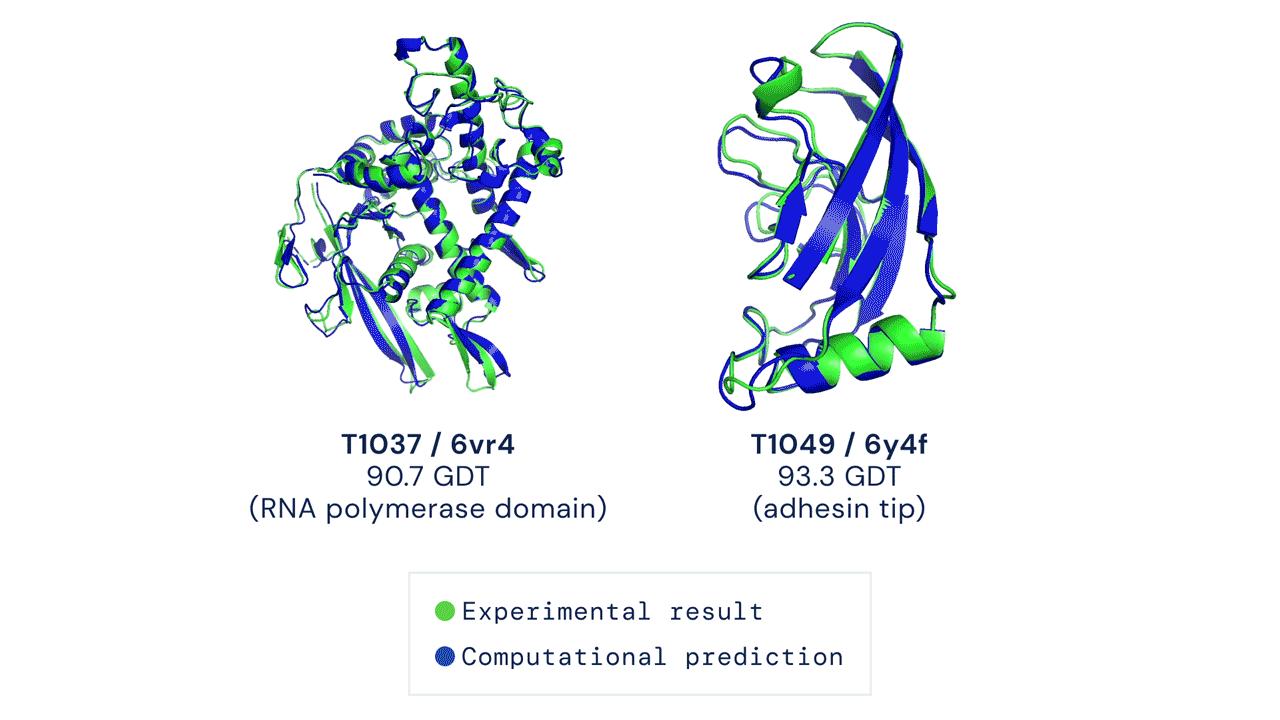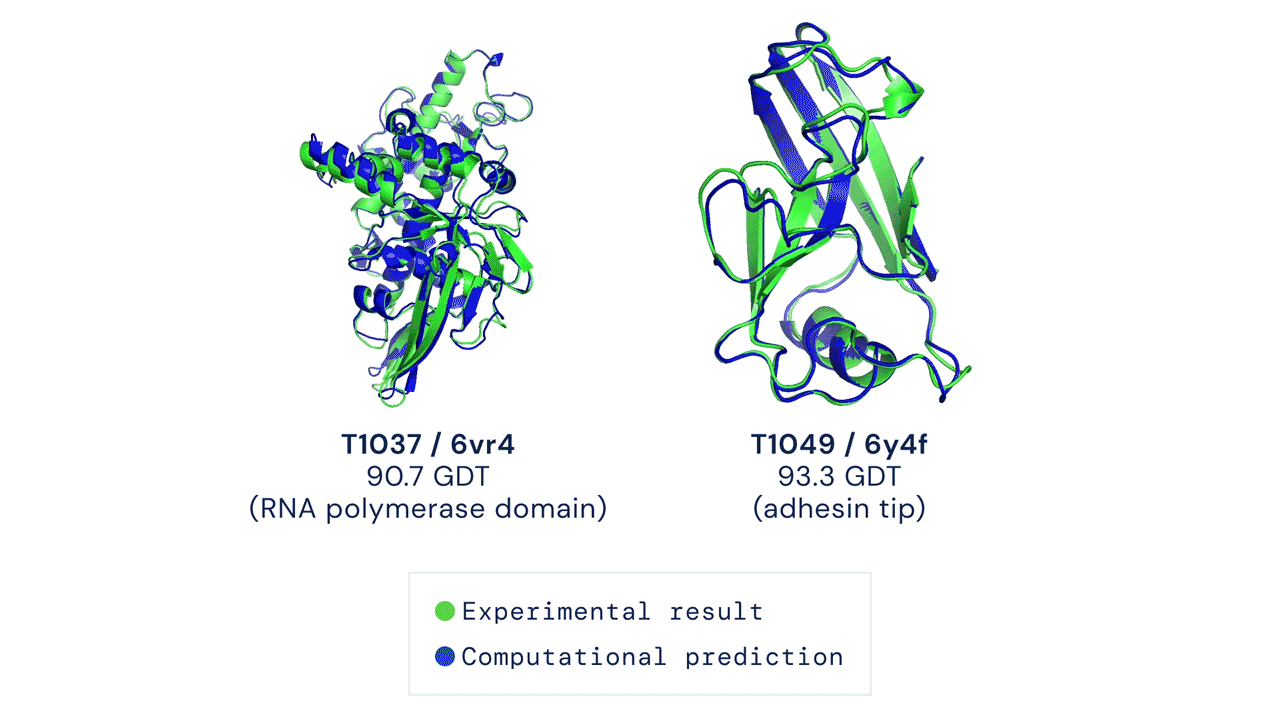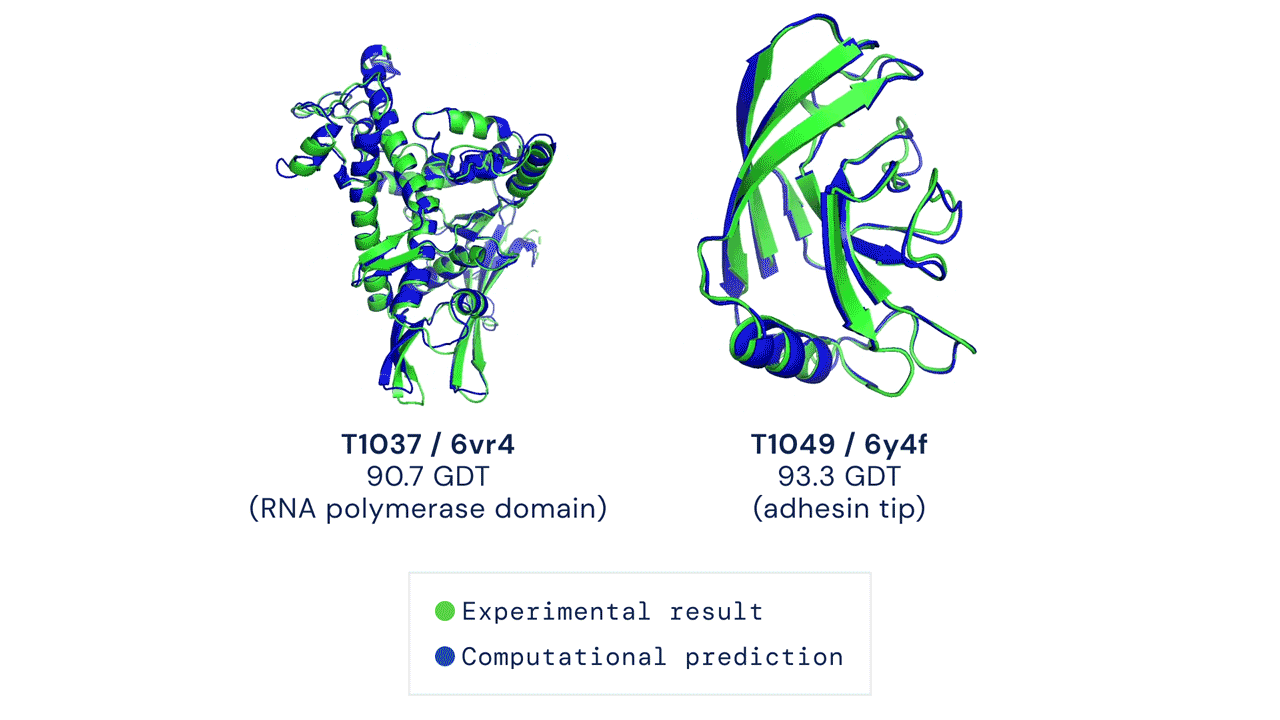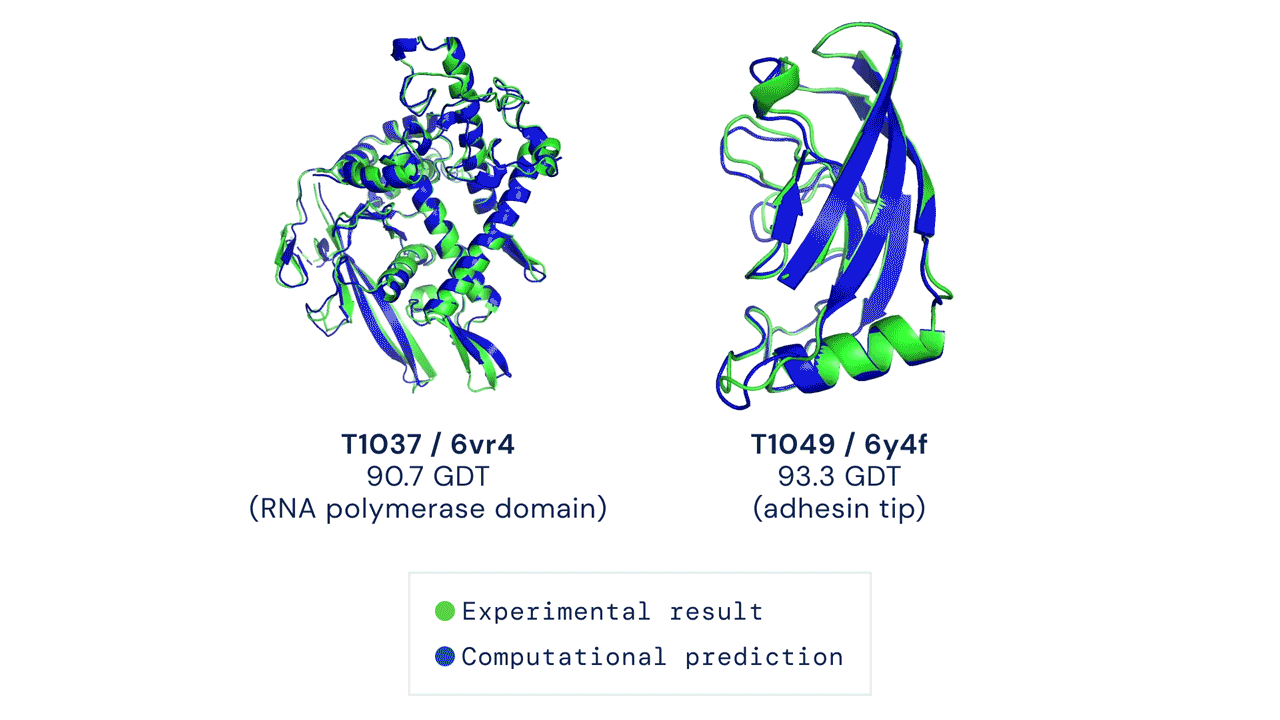
A szintetikus biológia korában élünk, de a mesterséges élet létrehozása még mindig csak álom

Ma már rendelkezésünkre áll a technológia baktériumok teljes genetikai információjának átírásához, emlősök, köztük az ember genomjának szerkesztéséhez vagy kórokozókat terjesztő élőlények kontrollálásához, miközben egyre jobban kezdjük megismerni az élet alapjául szolgáló genetikai rendszerek és sejtek működését is.
Nem kérdés tehát, hogy a szintetikus biológia korát éljük, és sosem voltunk még ilyen közel ahhoz, hogy új élőlényeket hozzunk létre laboratóriumban, vagy éppen különböző betegségeket kezeljünk hatásos génterápiával. Ugyanakkor a tudományos és technológiai áttörések az elmúlt 10–20 évben súlyos etikai kérdéseket is felszínre hoztak, talán legerőteljesebben az emberi genom örökölhető tulajdonságainak megváltoztatása körül.
Biohacking: világító növényektől bioszenzorokig
A szintetikus biológiáról a Jurassic Park genomszerkesztéssel létrehozott dinoszauruszai vagy a disztópikus biopunk jövőt megjelenítő Gattaca után leginkább a világító baktériumok és növények juthatnak eszünkbe. Ilyeneket viszonylag könnyen létre lehet hozni komplex laboratóriumi eljárások nélkül, így közkedveltnek számítanak a csináld magad biológiával (DIYbio) vagy biohackinggel foglalkozók körében.
A világító élőlényeket elsősorban a zöld fluoreszcens fehérjének (GFP) és különböző színű változatainak köszönhetjük, amiket molekuláris biológiai kutatásokban elterjedten alkalmaznak a génkifejeződés jelzésére. A fehérjét, mely kék vagy ultraibolya fény hatására világoszölden fluoreszkál, eredetileg az Aequorea victoria medúzából izolálta Simomura Oszamu szerves kémikus és tengerbiológus, valamint kollégái, akiket 2008-ban kémiai Nobel-díjjal ismertek el felfedezésükért.

A GFP-t vagy a sárga színű YFP változatát kódoló gént szakemberek egy kutatóintézetben, illetve biohackerek egy közösségi laborban rekombináns DNS technikával tudják bejuttatni egy baktérium, állat, vagy növény genomjába, ahol azokat a sejt vagy sejtek ki tudják fejezni. Ezek nem csak kutatási segédletek vagy művészi látványosságok lehetnek, hanem a biológiai szenzorok létrehozását is megalapozzák. Ilyen a Pseudomonas fluorescens HK44 baktérium is, amely petróleumszennyeződések jelzésére képes, de a fluoreszcens fehérjéket felhasználó bioszenzorok az élőlények állapotáról, vagy megbetegedésük státuszáról is árulkodhatnak.
A biohacking céljairól, illetve a műveléséhez szükséges, elérhető árú molekuláris biológiai műszerekről Sebestyén Endre bioinformatikus beszélt hosszabban a 2019-es Qubit live-on tartott előadásában. Előadásából az is kiderül, hogy a csináld magad biológia a génexpressziót szabályozó, legószerű DNS-darabkák és a genomszerkesztés elérhetőbbé válásával már a közeljövőben jelentősen túlléphet a világító életformák létrehozásán.
Technológiai forradalmak ágyaznak meg a szintetikus biológiának
Az elmúlt 20 évben több olyan technológia sokkal megfizethetőbbé, hatékonyabbá és pontosabbá vált, amely kulcsfontosságú a szintetikus biológiához, tágabb értelemben pedig a genetikai és molekuláris biológiai kutatásokhoz.
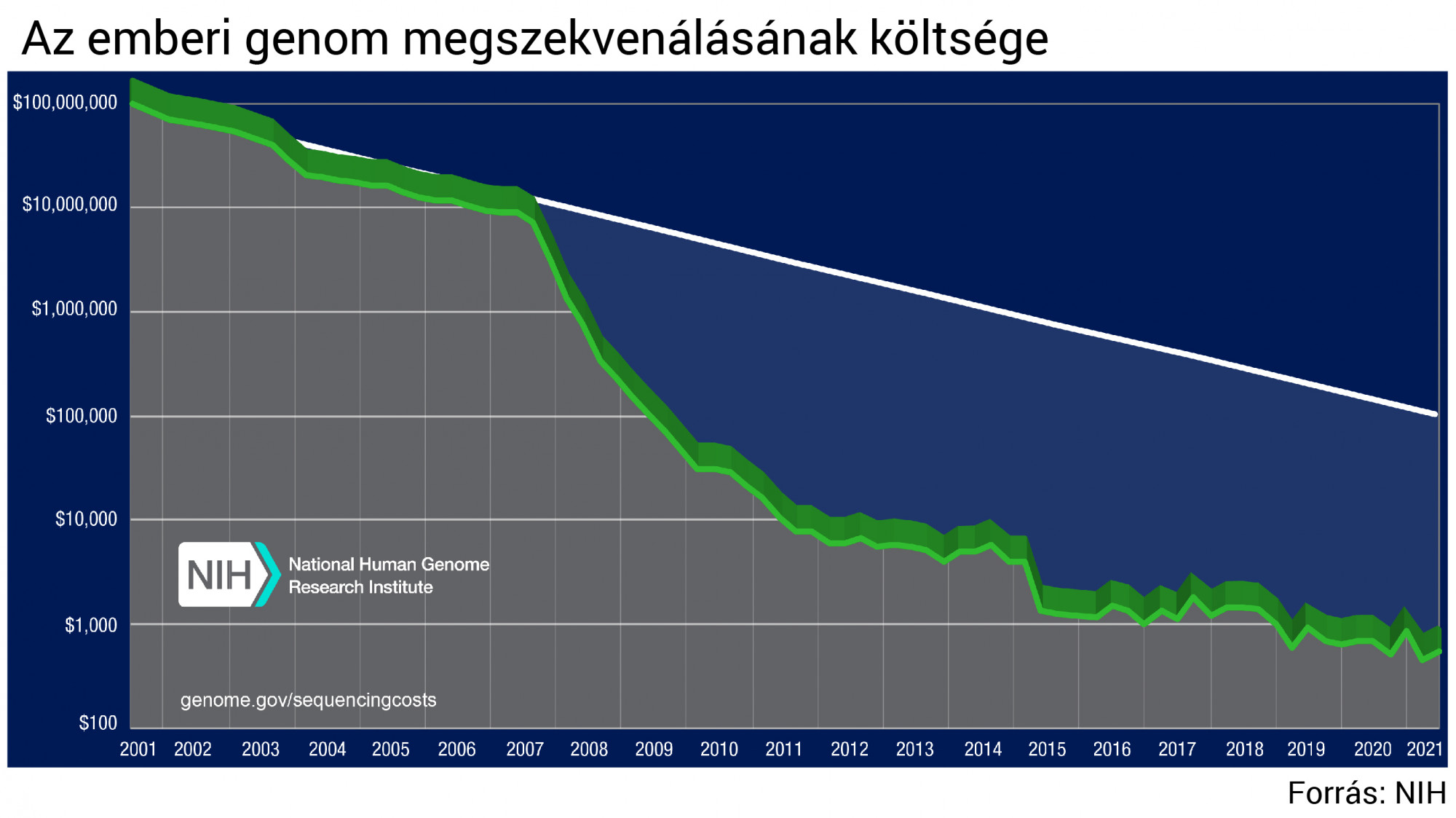
A legfontosabb ezek közül az élőlények DNS-bázissorendjének meghatározására szolgáló szekvenálás, amelynek költsége az elmúlt húsz évben meredeken zuhant, miközben egyre nagyobb genomok felderítése vált lehetségessé, egyre gyorsabban. Az amerikai egészségügyi intézet (NIH) adatai szerint 2001-ben még majdnem 100 millió dollár volt egy emberi genom szekvenálási költsége, ami 2021-re 1000 dollár környékére zuhant. Ehhez nagyrészt az újgenerációs szekvenálási technológiák (NGS) megjelenése vezetett, melyek párhuzamosan képesek rengeteg apró DNS-szakasz (short read) bázissorendjének feltárására, és ha azokat számítási módszerekkel összeillesztik, egy élőlény teljes genetikai információja rekonstruálható. Kutatók közben már harmadik generációs szekvenálási technológiákon is dolgoznak, melyek az NGS módszereknél hosszabb szakaszokat (long read) képesek megszekvenálni, ami csökkenti a hibák lehetőségét.
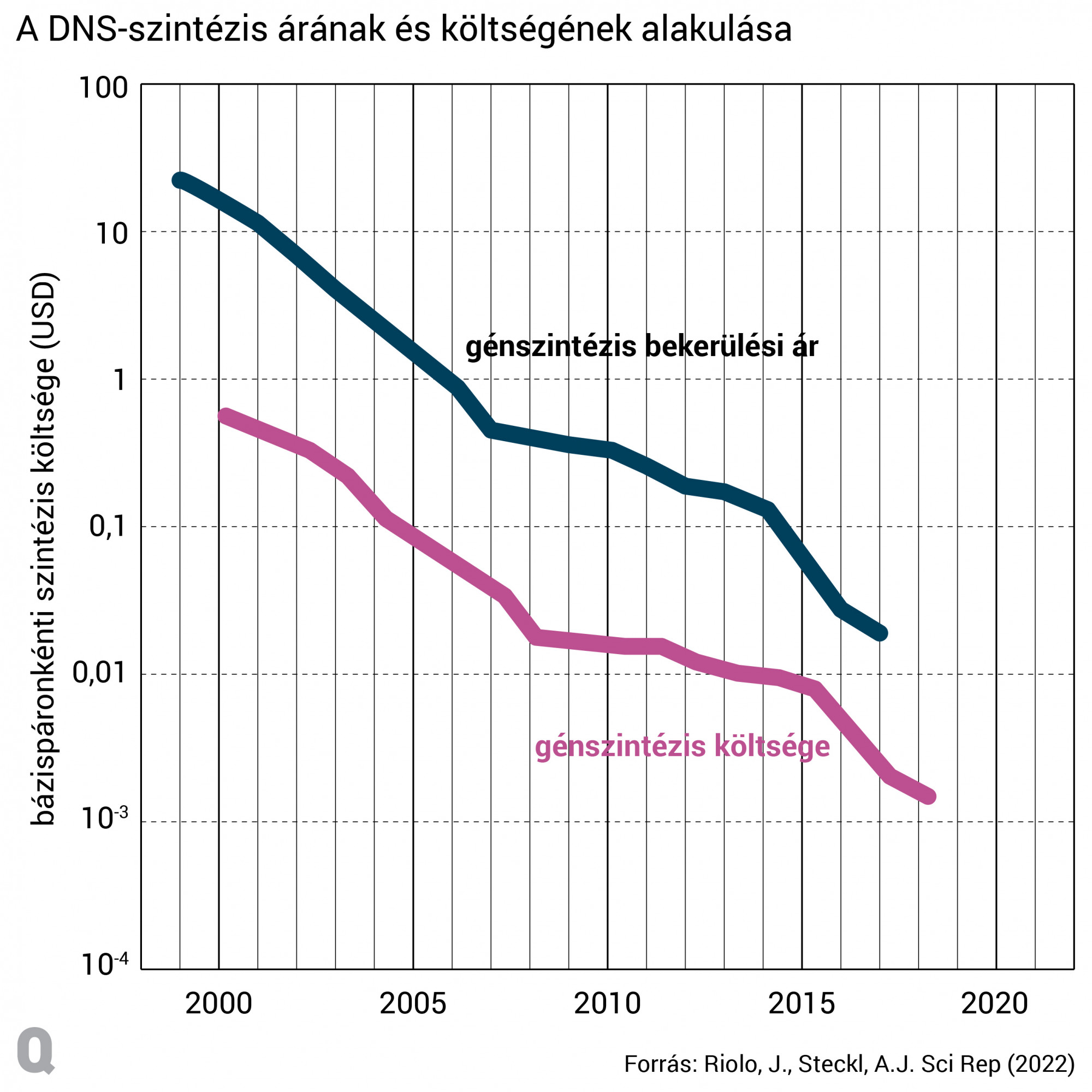
A DNS-kód meghatározása mellett a DNS-szintézis, vagyis a gének és más DNS-szekvenciák laboratóriumi létrehozása is sokkal megfizethetőbbé vált az elmúlt néhány évben. A technológia, melynek eredete az 1970-es évekre nyúlik vissza, legtöbbször 200 bázispárból álló oligonukleotidok kémiai úton történő létrehozásából és a darabkák összeillesztéséből tevődik össze. Egy idén februárban, a Scientific Reportsban közölt tanulmány szerint egy gén szintetikus megalkotásának költsége és piaci ára is meredeken zuhant az elmúlt két évtizedben. Míg az ezzel foglalkozó cégek 2000-ben bázispáronként több mint 10 dollárt számoltak fel (egy humán gén teljes hosszúsága átlagosan 67 ezer bázispár), manapság ez inkább 0,05 dollár körül alakul, vagy még annál is olcsóbb.
A DNS-szekvenáláson és -szintézisen túl a szintetikus biológia arzenáljának megkerülhetetlen része a gén- és genomszerkesztés is, amikor a DNS egy része törlésre, illetve módosításra kerül, vagy egy új DNS-szakaszt illesztenek be az élőlény genetikai kódjába. A korábbi, kevésbé hatékony és lassabb megoldásokat a 2010-es évek közepén egy áttörő módszer, a CRISPR váltotta fel. Ezt a sejtmag nélküli (prokarióta) mikrobák, az archeák és a baktériumok által fág vírusok elleni védekezésre használt molekuláris rendszert több évtizednyi kutatás után, a 2000-es évek végén írták le.
A CRISPR (Clustered Regularly Interspaced Short Palindromic Repeats) olyan ismétlődő és a sejt molekuláris gépezete által mindkét irányból olvasható DNS-szekvenciákat takar, melyek a mikrobát korábban megfertőző fág vírusokból származnak. Az ismert baktériumgenomok mintegy 50 százaléka, míg az archeák genomjának 90 százaléka tartalmaz CRISPR-szekvenciákat, melyeket későbbi vírusos fertőzésekkel szemben tudnak használni. A szekvenciák mellett a rendszer része a Cas9 enzim, amely a CRISPR DNS-t templátként használja fel, és így irányítja az ahhoz hasonló DNS-szálak felismerését és vágását.
2012-ben Jennifer Doudnának és Emmanuelle Charpentiernek hála kiderült, hogy a CRISPR-t RNS segítségével a gyakorlatban is lehet alkalmazni genomok szerkesztésére – a kutatóknak ezért az áttörő felfedezésért 2020-ban ítélték meg a kémiai Nobel-díjat. A 2010-es évek közepére az ő és más kutatók erőfeszítéseinek köszönhetően a CRISPR már a genetikai és szintetikus biológiai kutatások meghatározó genomszerkesztési módszerévé vált. A CRISPR-t ezen túl nem csak genom szerkesztésre, hanem élőlények génexpressziójának szabályozására is lehet használni, illetve olyan viselhető bioszenzorok létrehozását is lehetővé teszi, amelyek vírusos fertőzéseket, így például a covidot okozó SARS-CoV-2-t is képesek kimutatni.
Idővel a mélytanuláson alapuló mesterséges intelligencia is fontos része lehet a szintetikus biológiai eszköztárnak. Amint arról beszámoltunk, két hónapja kutatók egy olyan algoritmust közöltek, amely képes a DNS-bázissorend alapján előre jelezni gének kifejeződésének intenzitását, legalábbis élesztőgombák esetén. De részletesen bemutattuk azt az Alphafold2 nevű algoritmust is, amely a fehérjék térszerkezetét azok aminosav-sorrendjéből minden korábbi számítási módszernél pontosabban képes megfejteni – ennek leírását tavaly közölték a Nature-ben, illetve nyíltan elérhetővé is tették az algoritmust a GitHubon.
CRISPR-babák és a gene drive-ok
Ahogy az új, forradalmi technológiákkal időnként előfordul, a CRISPR felhasználásának is gyorsan megjelentek az árnyoldalai. Erre kevés erősebb példa van, mint Ho Csian-kuj kínai kutató esete, aki örökölhető változtatásokat eszközölt a CRISPR segítségével egy ikerpár, valamint egy harmadik gyermek genomjában. Ho Csian-kuj 2018-as bejelentése az ikerpár CCR5 génjeinek örökölhető módosításáról, amit a kutató az AIDS-et okozó HIV-vírussal szembeni (komolyan vitatható) rezisztencia létrehozásával indokolt, hatalmas nemzetközi botrányt kavart. A kutatót 2019 decemberében Kínában 430 ezer dollárra bírságolták meg, és 3 év börtönbüntetésre ítélték, amit nemrég fejezett be.
A kínai kutató tettének következményeiről Varga Máté, az ELTE TTK Genetikai tanszékének docense a Qubit emailben küldött kérdésére elmondta, hogy „magukra adó kutatók korábban sem tekintették sehol realisztikusnak az ivarvonalvonal-szerkesztést a közeljövőben, és ez azóta még inkább így lett, hiszen ez az egész egy nagyon hosszú és sötét árnyat vet minden ilyen típusú törekvésre”. A Qubiten is rendszeresen publikáló genetikus 2019-ben, a Qubit live-on beszélt arról, hogy az etikai problémák mellett jelenleg szinte képtelenség a komplex tulajdonságok célzott megváltoztatása, mivel ahhoz akár több ezer vagy tízezer helyen kellene a genomot precízen módosítani, és még ennek sem lenne garantált az eredménye.
A genomszerkesztés nem csak akkor vezethet nagy vitákhoz, ha emberekről van szó. Erre a legjobb példát az úgynevezett gene drive-ok jelentik, amelyek olyan, mesterségesen a genomba illesztett gének, melyek rövid idő alatt maguktól elterjednek egy adott populációban, mivel a normál öröklődéshez képest nagyobb valószínűséggel adódnak át az utódnemzedéknek – a gene drive-okhoz képest a növénytermesztésben alkalmazott genetikailag módosított organizmusok (GMO) megváltoztatott genetikai szakaszai nem képesek az önmaguktól való terjedésre.
Támogatóik szerint a gene drive-ok lehetővé tehetik a tömeges emberi megbetegedésekért felelős gazdaállatok, például a maláriát terjesztő szúnyogok populációinak genetikai módosítását, kontrollálását, vagy extrém esetben megszüntetését, amivel éves szinten akár több millió életet lehetne megmenteni. Ellenzői szerint viszont a gene drive-ok bevetése előre nem látható következményekkel járhat, míg az amerikai szövetségi nyomozóiroda (FBI) néhány éve amiatt fejezte ki az aggodalmát, hogy a technológiát bioterrorizmusra is fel lehet használni.
Varga szerint – főleg a szúnyogok esetében – ígéretes laboratóriumi eredményeket értek már el gene drive-okkal, de népszerűek a nem gene drive-okat alkalmazó megoldások is, például a szúnyogokban természetesen is megtalálható, és különböző vírusok, például a nyugat-nílusi vírus ellen számukra nagyobb rezisztenciát biztosító Wolbachia baktériumokkal fertőzött egyedek kiengedése a természetbe. Közben a gene drive-ok biztonságosabbá tételére és kontrollálhatóságára is vannak törekvések, olyan, CRISPR-rendszert alkalmazó megoldásokkkal, melyek laboratóriumi tesztekben képesek voltak a gene drive-ok terjedését megállítani egy populációban.
Nagyon messze vagyunk az első teljesen mesterséges sejtektől
Egy genom néhány helyen történő szerkesztéséhez képest egészen más kihívás egy teljes genom újonnani létrehozása, nem beszélve egy kizárólag laboratóriumban megalkotott komponensekből összerakott mesterséges sejtről. Az erre törekvő kutatások potenciálisan segíthetnek az élet eredetének megértésében, vagy különböző biotechnológiai alkalmazásokkal rendelkező új életformákhoz vezethetnek el.
Az első szintetizált genommal rendelkező sejtet Craig Venter amerikai genetikus vezetésével, Daniel Gibson és kollégái hozták létre, akik 2010-ben a Science-ben publikálták eredményeiket. A kutatók egy Mycoplasma mycoides baktérium korábban nagy pontossággal megszekvenált genomján végeztek apró módosításokat digitálisan – nem esszenciális géneket töröltek, és úgynevezett vízjegyszekvenciákat helyeztek el. Ezután szintetizálták, majd összeállították azt, és elhelyezték a Mycoplasma capricolum sejtjeibe, melyeket utána már a szintetikus genom DNS-e irányított. Az új, Mycoplasma mycoides-ként viselkedő baktérium ezután a várt jellemzőket (fenotípus) mutatta, és képes maradt az önreplikációra.
Négy évvel később Flyod Romesberg és munkatársai még nagyobb lépést tettek: természetben nem előforduló DNS-bázisokat felhasználó plazmidot hoztak létre, és egy Escherichia coli baktérium genetikai információját egészítették ki egy kibővített genetikai kódszótárral. 2019-ben aztán Jason Chin és kollégái a Nature-ben közölték az első teljesen átkódolt, szintetizált genommal rendelkező E. coli-t, amely átírt kódszótárjával, illetve egy korábban a fehérjeszintézishez létfontosságú transzfer RNS nélkül is életben tudott maradni.

Varga úgy látja, ezek a kutatások a nagyméretű genomszintézis szempontjából mindenképpen előrelépést jelentettek, ugyanakkor még mindig nem tudunk teljesen a semmiből (de novo) genomot tervezni. Ennek szerinte az az alapvető oka, hogy a kísérletekben használt baktériumok minimálgenomjai is tartalmaznak meglepetéseket, és olyan fehérjéket, szabályozó DNS- és RNS-motívumokat, amelyeknek nem értjük a valódi funkcióját.
Érdemes még itt kitérni Sebestyén Endre egy 2018-as, a Budapest Science Meetupon tartott előadására, amelyben a kutató arra hívta fel a figyelmet, hogy a természetes sejtek normál esetben is tele vannak zajos és hibázó mechanizmusokkal, hiszen 4 milliárd évnyi evolúció eredményeiről, és nem precíz gépekről van szó. Az ilyen kísérletek így valódi mesterséges élet létrehozása helyett a gyakorlatban inkább a lehető legkisebb funkcionális genommal elműködő élőlény létrehozására vállalkoztak, és nem egy teljesen szintetikus sejtére, ahol a molekuláris apparátus és a környezettől a sejtet elhatároló membrán létrehozásához sem rendelkezünk megfelelő ismeretekkel vagy technológiával.
Mint Varga a Qubitnek elmondta, belátható időn belül csak úgy tudnánk egy teljes sejtet összerakni laboratóriumban, ahogy azt Craig Venterék tették, azaz kellene hozzá egy „béranya” sejt, amely biztosítaná, hogy a sejtbe vitt genom elkezd működni és replikálódni.
Kapcsolódó cikkek a Qubiten:

Hol tart ma a klónozás, mire jó a génszerkesztés, és mire képesek a biohackerek? Ilyen volt a Qubit Live #2
A DNS-forradalomról szólt a Qubit második élő estje, ahol öt neves kutató mutatta be, hol tart a tudomány a genetika különböző területein, többek között a rákkutatásban, a genom feltárásában vagy az antibiotikum-rezisztancia legyűrésében.

Ha az élet keletkezéséről van szó, a legkreatívabb fantázia sem tud versenyre kelni több százmillió év kémiai kísérleteivel
Az evolúciós átmenetek néven ismertté vált elméletet továbbfejlesztő budapesti iskola tagjai az elmúlt három évtized hazai eredményeit foglalták össze a Nature Reviews Chemistry-ben megjelent cikkükben. A tanulmány társszerzőjét, Szilágyi András biofizikust kérdeztük az élet eredetét firtató kutatásokról.

Tudományos dráma a génszerkesztés körül: miért ez a két tudós kapott Nobel-díjat a CRISPR/Cas9 feltalálásáért?
Egyszer még filmre vihetik a legreménytelibb génszerkesztési eljárás történetét, amelyben nagy egyetemek és tőkeerős cégek feszülnek egymásnak a szabadalomért egészen a mai napig. Az eljárás kidolgozásáért többen igényt tarthattak volna a Nobel-díjra, Emmanuelle Charpentier és Jennifer Doudna mégis megérdemelten vitte el a pálmát.
Kapcsolódó cikkek