Nem kell pánikba esni, ha nincs DNS-mintánk: új módszerekkel molekulák nélkül is jól feltárható a kihalt élőlények evolúciója

Genetikai információ segítségével pontosabban meghatározhatók az élőlények evolúciós kapcsolatai, mint az anatómiájuk alapján, mutat rá egy friss kutatás, amelynek eredményei igazolják az elmúlt évtizedekben a klasszikus rendszertan molekuláris módszerekkel való felülvizsgálatáért tett erőfeszítéseket.
Jack Oyston, az angliai Bath-i Egyetem evolúcióbiológusa és kollégái május végén, a Nature Communications Biology folyóiratban tették közzé eredményeiket, melyek szerint a molekuláris adatokon nyugvó, evolúciós törzsfákat létrehozó filogenetikai vizsgálatok pontosabban visszaadják az élőlények valós földrajzi elterjedését. Ezt a módszert viszont fosszíliák esetén, ahol általában semmilyen molekuláris információ nem őrződik meg, nem lehet bevetni, és ez felveti a kérdést, hogy milyen megoldások segíthetnek az őslények evolúciós rokonságának lehető legprecízebb feltárásában.
Ennek megválaszolásához két magyar paleontológust – Ősi Attilát, az ELTE TTK Őslénytani Tanszékének vezetőjét és Rabi Mártont, a Tübingeni Egyetem Őslénytani tanszékének kutatóját – kerestünk meg, akik korábban végeztek, vagy jelenleg is végeznek fosszíliák morfológiai jellemzőin alapuló filogenetikai vizsgálatokat, amelyek rávilágítanak különböző élőlénycsoportok evolúciós történetére.
Általában jobb a molekuláris törzsfa, de azért nem mindig
A 20. század második felének molekuláris biológiai forradalma lehetővé tette élőlények genetikai információjának, fehérjéik aminosavsorrendjének, RNS-molekuláik, valamint az összes sejtes élőlénynél örökítőanyagul szolgáló DNS-ük bázissorendjének megismerését. 1977-ben Carl Woese és George Fox riboszomális RNS-szekvenciák vizsgálatával ért el fontos fontos áttörést, amikor azonosították az archeákat, vagyis a baktériumok és az eukarióták után a földi élet harmadik nagy csoportját, amellyel korábban már a Qubiten is részletesen foglalkoztunk.
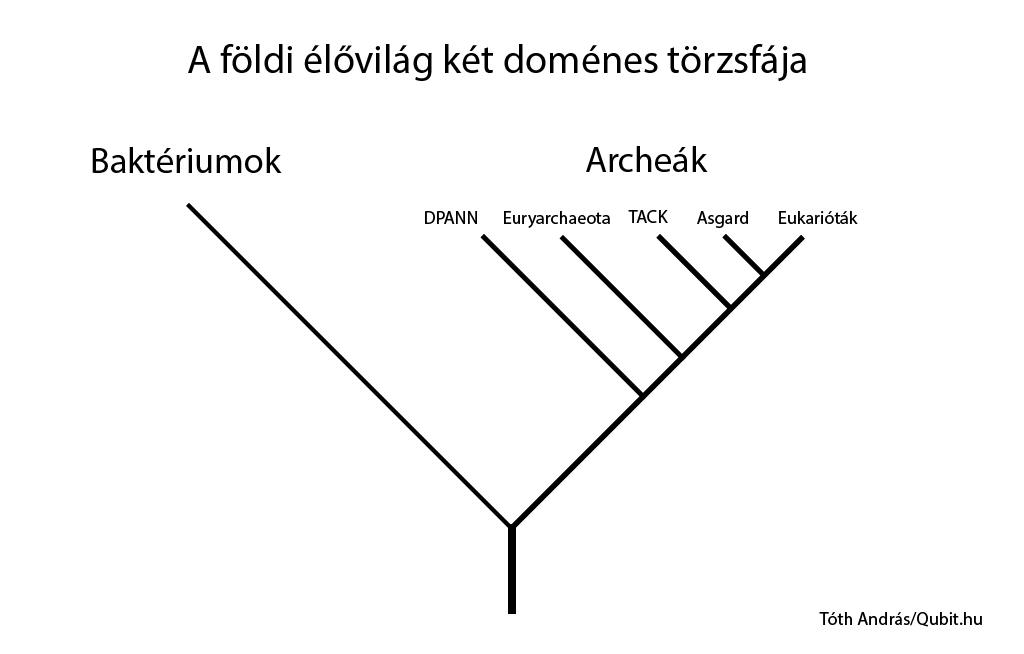
Az 1990-es évektől az első baktérium-, majd állati genomok szekvenálásával nagy lendületet kapott a molekuláris filogenetika, és hol kisebb, hol nagyobb módosításokkal átíródott az élet törzsfája. Oystonék szerint erre jó példát jelentenek a méhlepényes emlősök, ahol a molekuláris fák által azonosított emlőscsoportok (Afrotheria, Atlantogenata, Boreoeutheria és Laurasiatheria) jobban visszaadták ezen emlősök mai földrajzi elterjedését. De egyes csoportoknál, mint az ízeltlábúak, hüllők, és madarak esetében úgy vélekednek, hogy a morfológiai és molekuláris alapú vizsgálatok jól kiegészítik egymást. A kutatók szerint már korábban is utaltak arra szimulációk, hogy a ma élő fajok esetén gyakran alkalmazott molekuláris módszerek jobb eredményt adnak, de eddig kevés statisztikai vizsgálat foglalkozott a kérdéssel.
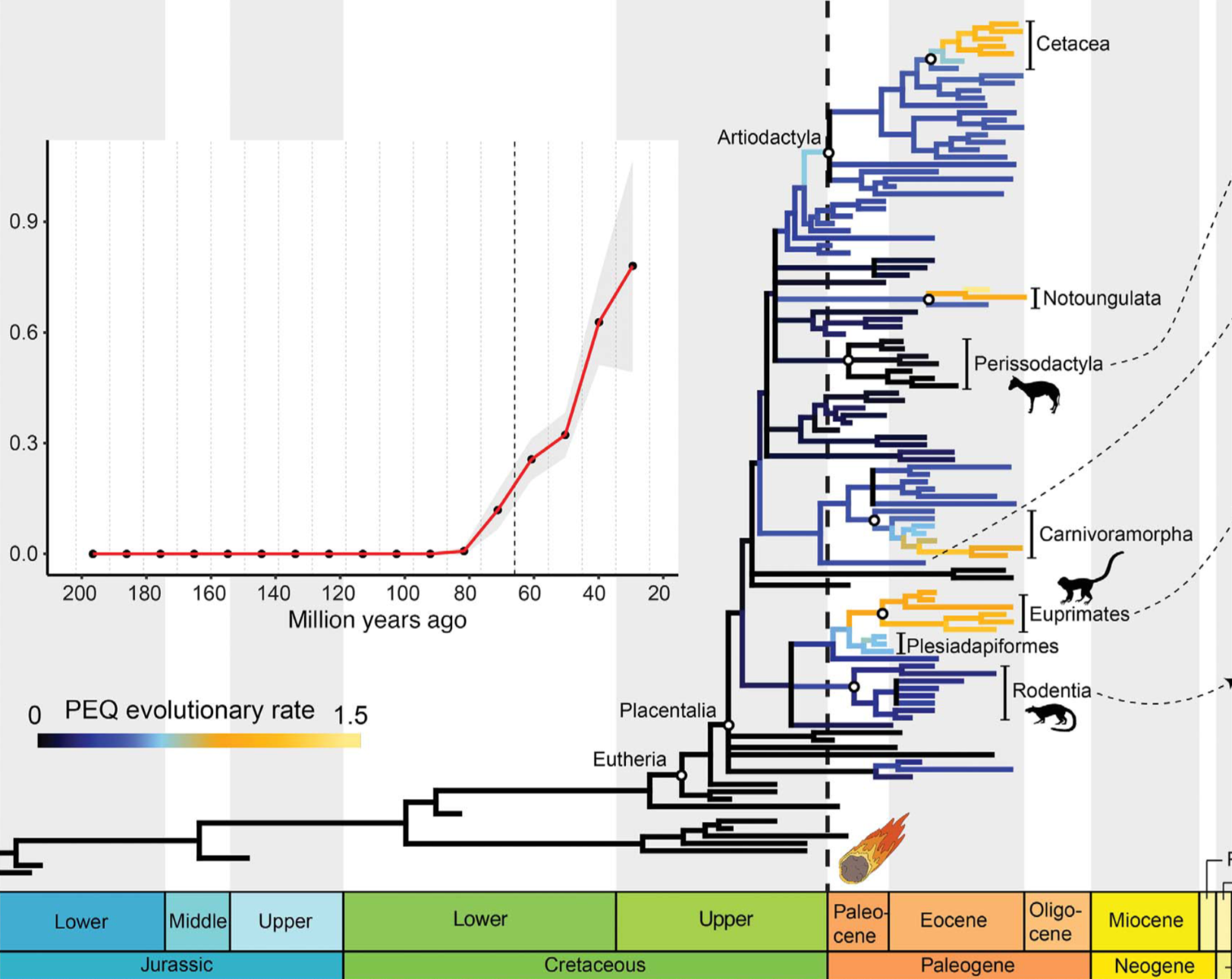
Oyston munkatársaival 48 pár molekuláris és morfológiai adatokon alapuló állati és növényi törzsfát hasonlított össze, melyeket 1981 és 2015 közt publikáltak, és pontosságukat két független kritériummal szemben határozta meg. Egyrészt azt nézték meg, hogy mennyire passzolnak a törzsfák az élőlények földrajzi elterjedéséhez (a Természetvédelmi Világszövetség által felállított és más adatbázisok alapján), ezután pedig a fosszilis rekordhoz, konkrétabban a vizsgált élőlényeknél az első fosszíliáik megjelenéséhez hasonlították őket.
A megvizsgált morfológiai törzsfák 54, míg a molekulárisak 65 százaléka jobban illeszkedett a földrajzi elterjedéshez, mint azt véletlenszerű adatoknál várnánk, és összességében a molekuláris fák jelentősen jobban teljesítettek a földrajzi elterjedést tekintve. Ehhez képest a kutatók nem találtak szignifikáns eltérést 23 molekuláris és morfológiai fa között, amikor a fosszilis rekorddal (sztratigráfia) hasonlították össze őket.
Kiderült az is, hogy a molekuláris és morfológiai fák földrajzi elterjedésre vetített pontossága az elmúlt években nőtt, melynek okát a kutatók a növekvő adatmennyiségben, a fejlődő vizsgálati módszerekben és a jobb evolúciós modellekben látják. A törzsfák pontosságának időbeli növekedési trendje azonban önmagában nem magyarázza, hogy miért jobbak a molekuláris fák a morfológiai fáknál a földrajzi elterjedés visszaadásában. Ráadásul a trend alól vannak kivételek is, például a denevérek, kenguruk, vagy fenyők esetében, ahol a morfológiai fák jobban teljesítettek a méréseik szerint.
A kutatók a tanulmányuk végén hangsúlyozzák, az eredmények nem azt sugallják, hogy nyugdíjazni kellene a morfológiai módszert, hiszen egyes esetekben az kritikus lehet az élőlények közti kapcsolatok feltárásában, és a kihalt élőlények rokonsági és evolúciós kapcsolatainak meghatározásához legtöbbször kizárólag ilyen adatok állnak rendelkezésünkre. Ugyanakkor statisztikai vizsgálatuk azt jelzi, hogy a konvergens evolúció által létrehozott, az élőlények közti alaktani vagy anatómiai hasonlóságok félrevezethetnek a valós evolúciós rokonság meghatározásában.
Mi akkor a megoldás az őslénytanban?
„Jó pár évvel ezelőtt mi is csináltunk filogenetikai elemzéseket” - mondta a Qubit kérdésére Ősi Attila. Mint elmagyarázta, az általuk használt módszer lényege ekkor az volt, hogy egészen egyszerűen a csontokon megfigyelhető morfológiai karaktereket kódolták – vagyis azt vizsgálták, hogy megvan, vagy nincs meg a csonton például egy adott bütyök vagy taréj, illetve hogy a csontok arányai hosszabbak-e, mint szélesebbek, vagy egy adott nyílás ovális vagy inkább kör alakú.
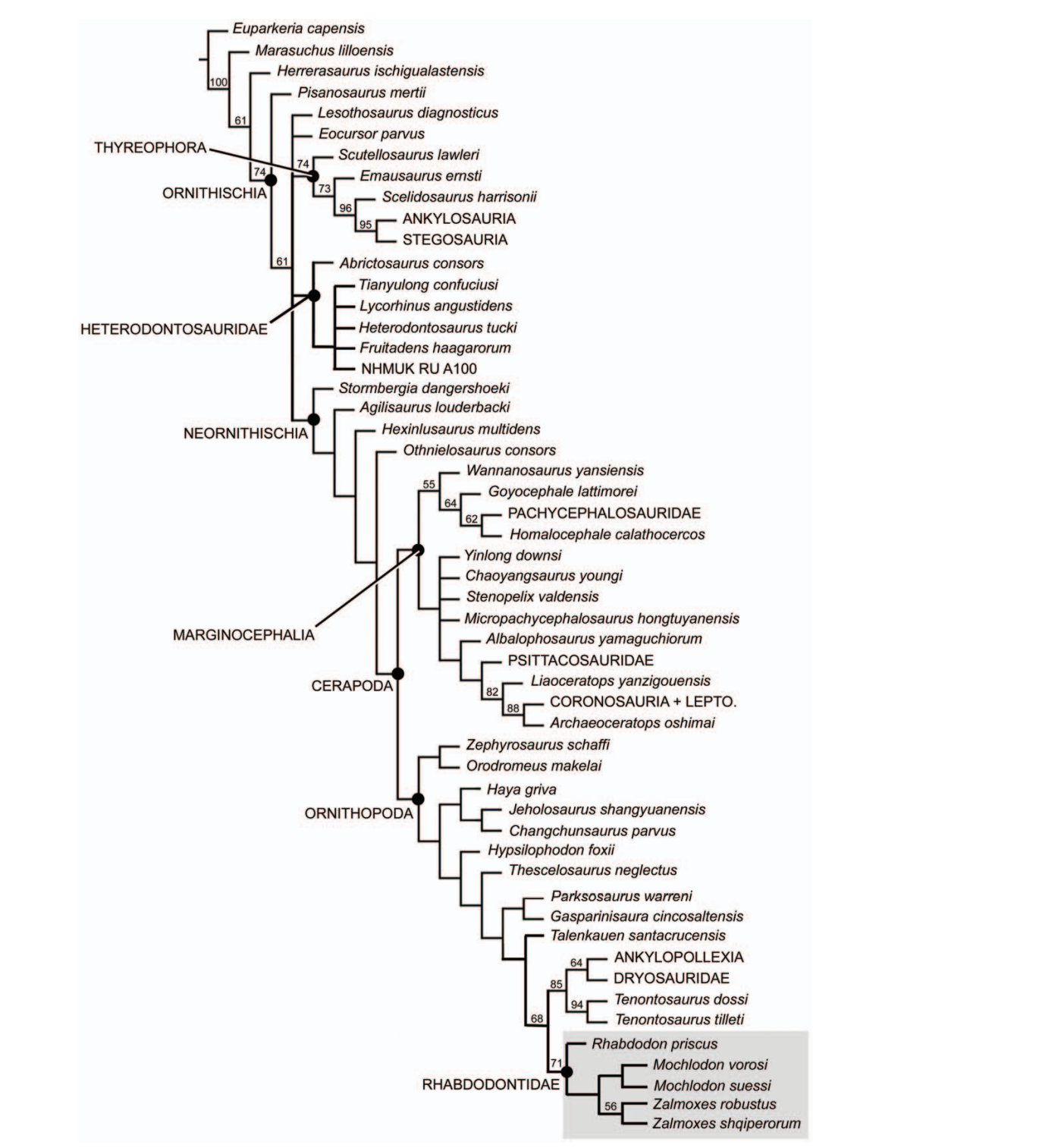
Rabi Márton, aki kollégáival a legkorszerűbb filogenetikai módszereket használja a krokodilok evolúciójának feltárására (tavaly közölt, a kajmánok észak-amerikai eredetére utaló kutatásukról itt írtunk), elmondta, hogy ha muszáj kizárólag morfológiai információkat felhasználva törzsfákat építeni, akkor egy úgynevezett tip-dating módszerhez érdemes folyamodni. Ezt Rabi és munkatársai nemrég alkalmazták is egy, a Biology Letters folyóiratban idén februárban közölt kutatásban, ahol a krokodilok korai evolúcióját – a morfológiai adatok mellett – ma élő állatokból származó molekuláris adatokkal írták le.
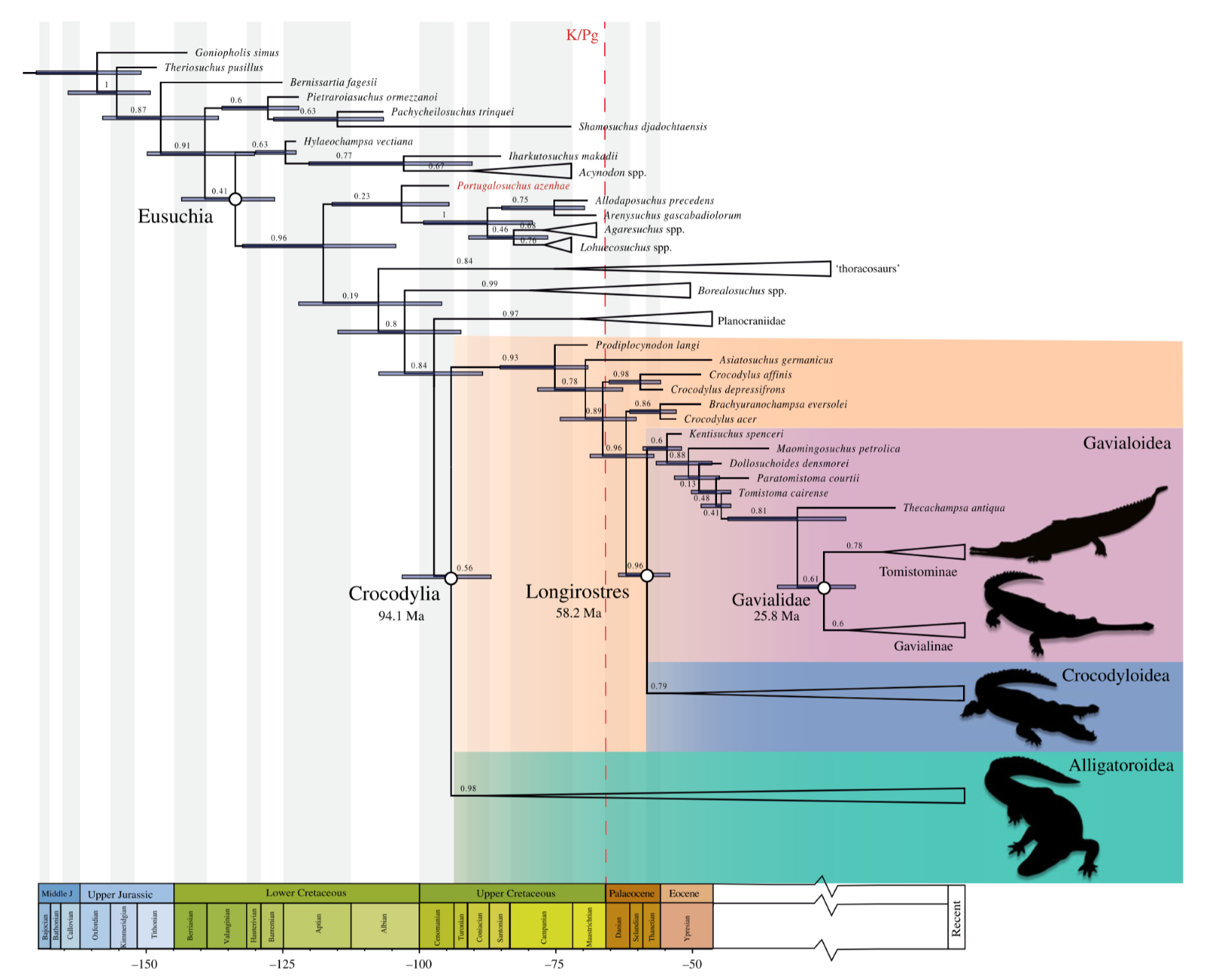
Rabi szerint a tip-dating egyfajta morfológiai óra (a DNS, RNS vagy fehérjék eltelt időre vetített átlagos mutációs rátáját megadó molekuláris óra analógiájára), melynek előnye, hogy a fosszíliák korát is számításba veszi a törzsfa rekonstrukciójánál. Mint a kutató elmondta, ezekből a módszer megbecsüli a morfológiai evolúció mértékét az egyes ágakon, és elkerüli a nagy ugrásokat.
Arra, hogy miért fontos ezeknek az ugrásoknak az elkerülése, Rabi úgy válaszolt, hogy a modell olyan topológiát és divergencia (az evolúciós ágak szétválására utaló) kort keres, ami konzisztens a fosszíliák korával, és az egyszerre számított morfológiai evolúciós rátával. A kutató szerint valójában ettől még nagy ugrások is lehetségesek, de csak akkor, ha ezt a fosszíliák jól dokumentálják.
Rabi úgy véli, hogy az egyszerű morfológiai filogenetikai vizsgálatok csak a legrövidebb, legkevesebb evolúciós lépéssel járó topológiát keresik, amelyek gyakran ellentmondanak a fosszíliák sztratigráfiai elterjedésének, azaz olyan evolúciót feltételeznek, amire valójában nincsen bizonyíték. Ehhez képest a kutató szerint a tip-dating módszerrel készült törzsfa ugyan egyes esetekben hosszabb lehet, erre az evolúciós mintázatra mégis több bizonyítékunk van fosszíliák formájában.
Kapcsolódó cikkek a Qubiten:

Észak-Amerikából származnak a kajmánok, derítette ki egy magyar szakember által vezetett kutatás
A Tübingeni Egyetem kutatója, Rabi Márton és munkatársai két különlegesen jó állapotban megmaradt fosszília alapján átírták mindazt, amit a kajmánok korai evolúciójáról tudunk.

113 millió éves toll írja át mindazt, amit eddig a tollak kialakulásáról gondoltunk
Most először írtak le a mai madarakéra emlékeztető színes tollakat, amiket a pteroszauruszok növesztettek. A rendkívüli felfedezés arra utal, hogy a tollaknak kezdetben hőszabályozási és vizuális kommunikációs szerepük volt – még jóval azelőtt, hogy a madárszerű dinoszauruszok szárnyra keltek volna.
A szintetikus biológia korában élünk, de a mesterséges élet létrehozása még mindig csak álom
Az elmúlt húsz év áttöréseinek köszönhetően mára lehetővé vált az élőlények, köztük az ember genomjának szerkesztése, de minden igyekezet ellenére még nagyon messze vagyunk attól, hogy teljesen szintetikus sejtet állítsunk elő.
