Az AlphaFold2 fehérjekutató algoritmus kifejlesztéséért és új fehérjék tervezéséért ítélték meg a 2024-es kémiai Nobel-díjat

A fehérjék háromdimenziós szerkezetének mesterséges intelligencia alapú meghatározásáért és új fehérjék tervezéséért ítélték meg a 2024-es kémiai Nobel-díjat. A díjat megosztva kapta David Baker, valamint Demis Hassabis és John Jumper.
Az indoklás szerint David Baker, a Washingtoni Egyetem biokémikusa véghezvitte a szinte lehetetlent, és laboratóriumában teljesen új típusú fehérjéket alkotott meg. Demis Hassabis, a Google DeepMind vezérigazgatója és John Jumper, a DeepMind egyik vezető AI-kutatója pedig az AlphaFold2 mesterséges intelligencia kifejlesztéséért részesült az elismerésben, hiszen az praktikusan megoldotta a fehérjék háromdimenziós szerkezetének meghatározását.
A fehérjék a földi élet nélkülözhetetlen szereplői, amelyek az életfolyamatokhoz szükséges reakciókat katalizálnak, és sejtbeli, valamint sejteken kívüli struktúrákat alkotnak. A makromolekulák általában 20-féle aminosav láncolatából épülhetnek fel, és egy fehérjefeltekeredésnek nevezett folyamat során nyerik el biológiailag funkcionális szerkezetüket. Ennek a folyamatnak a megértése évtizedekig a molekuláris biológia egyik legnagyobb megoldatlan problémája volt, és egy-egy fehérje szerkezetét csak nagyon költséges kísérleti módszerekkel lehetett megállapítani.

Baker 1962-ben született Seattle-ben, majd a Kaliforniai Egyetemen (Berkeley) doktorált. A Washingtoni Egyetem biokémikusa a Rosetta fehérjeszerkezet meghatározó algoritmus kifejlesztéséről is ismert, de a megosztott Nobel-díjat 2003-as, a Science folyóiratban közölt munkájáért kapta, amikor kollégáival aminosavakból mesterséges, egy addig a természetben sosem látott szerkezetű fehérjét hozott létre.
A 62 éves biokémikus kutatócsoportja, a Baker Lab jelenleg elsősorban fehérjetervező szoftverek kifejlesztésével foglalkozik, amiket új fehérjemolekulák megalkotására használnak. A szakemberek az algoritmusokkal olyan új fehérjéket terveznek, amelyek a jövőben gyógyszer-hatóanyagokként, vakcinák komponenseiként vagy akár nanoméretű szenzorokként szolgálhatnak. Baker és kollégái legutóbb 2022-ben a ProteinMPNN mélytanulási algoritmust mutatták be. Ez, mint megírtuk, állításuk szerint jelentősen gyorsabb a korábbi módszereknél, és nagyobb eséllyel ad ki működő molekulákat, ami megkönnyítheti a kutatóknak új, terápiás hatású fehérjék és nanostruktúrák létrehozását.
Hassabis és Jumper az 2020-as CASP14 fehérjealgoritmus-versenyen debütáló AlphaFold2 megalkotásában játszottak úttörő szerepet. A 2021-ben a Nature folyóiratban bemutatott algoritmus segítségével azóta lényegében az összes ismert – közel 200 millió – fehérje háromdimenziós szerkezetét sikerült meghatározni. Hassabis 1976-ban született Londonban, és 2010-ben alapította meg az akkor gépi tanulással foglalkozó startupként induló DeepMindot Shane Legggel és Mustafa Suleymannal.
A Google által 2014-ben felvásárolt startup eddigi legnagyobb dobásai az AlphaFold mellett a Go játékban a legjobb emberi játékosokat is legyőző AlphaGo és a középtávú időjárás előrejelzésre használható GraphCast algoritmusok voltak. A most 48 éves Hassabis azóta is a DeepMind vezetője, és nemrég megalapította a kifejezetten gyógyszerhatóanyagok kifejlesztését célzó startupját, az Isomorphic Labset. Jumper 1985-ben született az arizónai Little Rockban, és doktori címét 2017-ben szerezte meg a Chicagói Egyetemen, majd csatlakozott a DeepMindhoz, és részt vett az AlphaFold kifejlesztésében. Emiatt 2021-ben a Nature folyóirat beválogatta azon 10 kutató közé, akik jelentősen formálták a tudományt abban az évben.
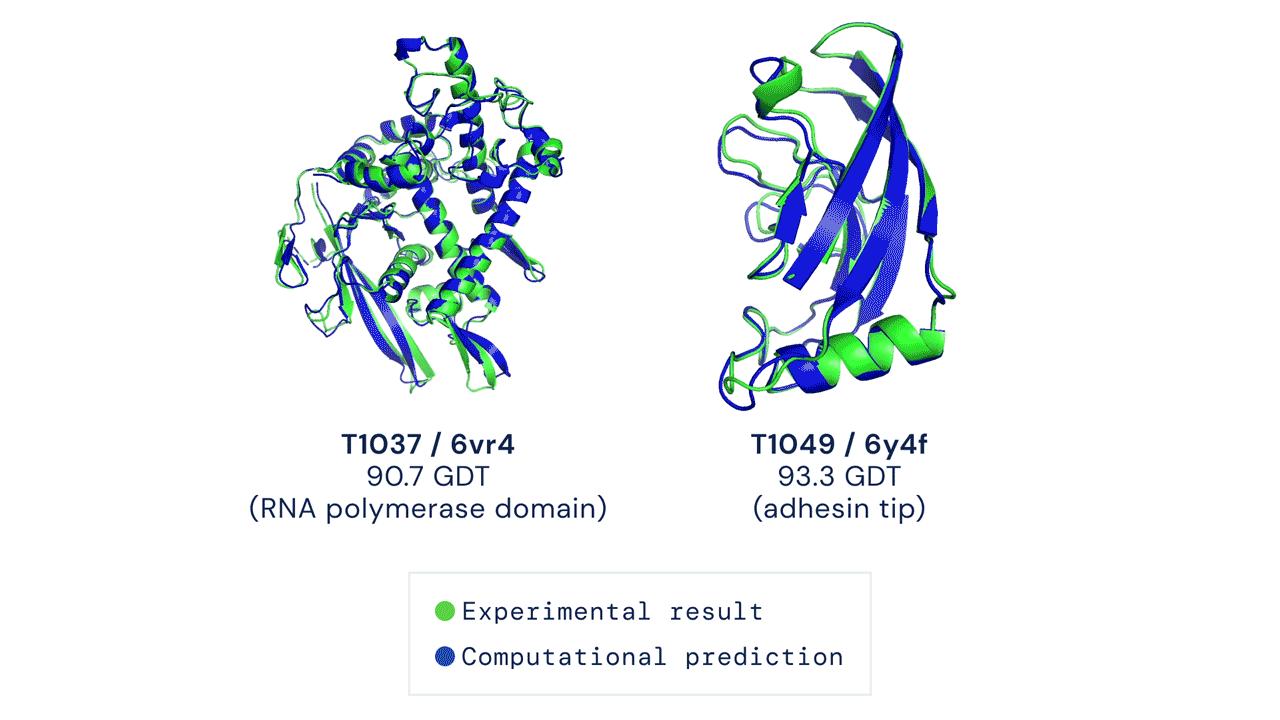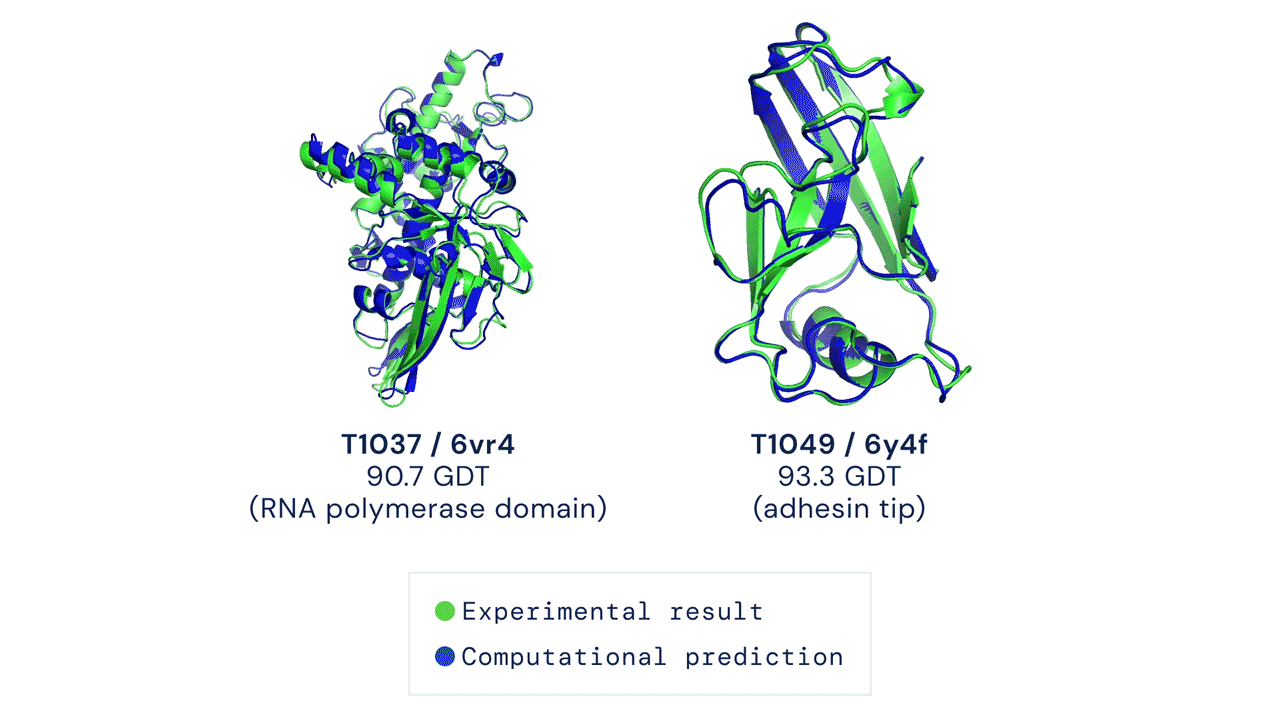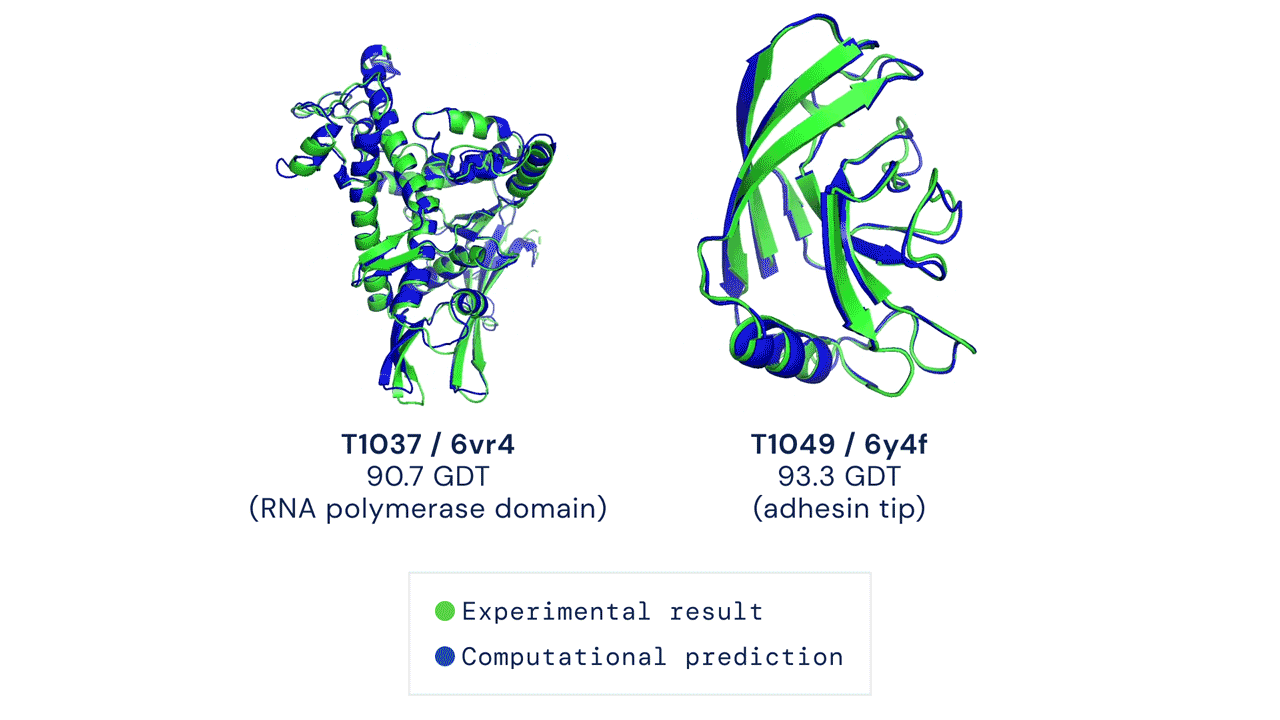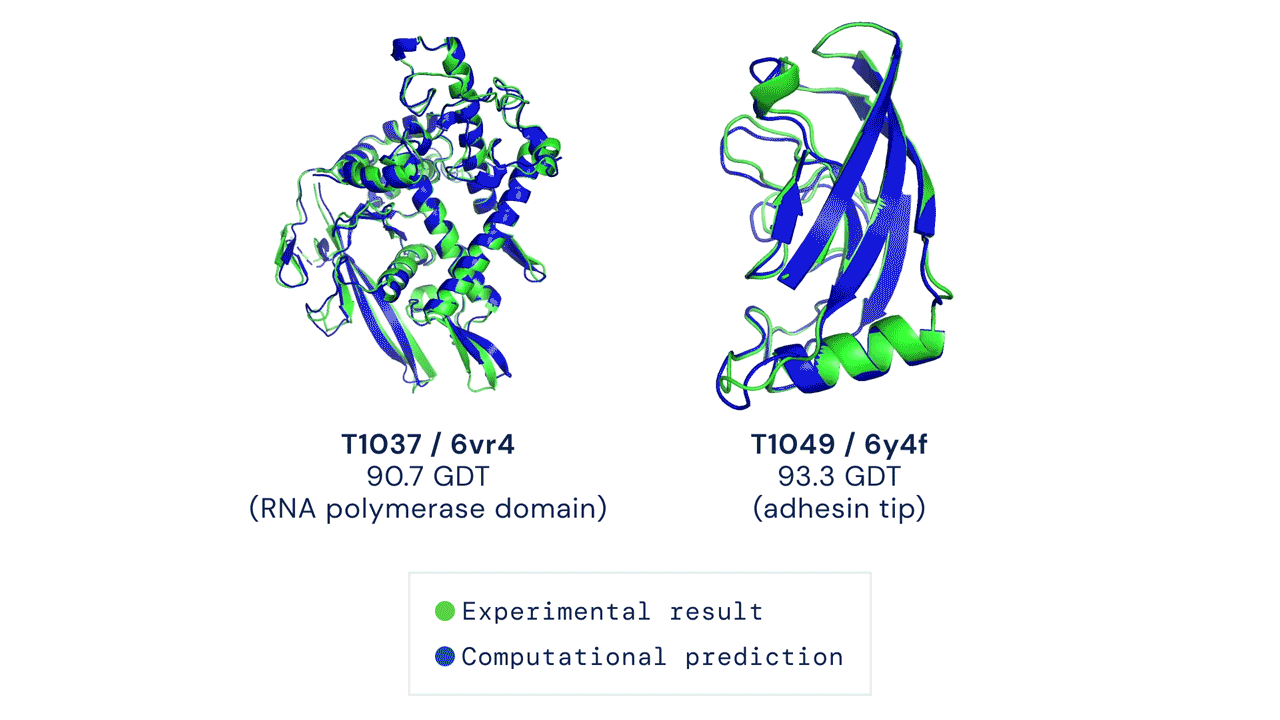
Több százmillió fehérje szerkezetét határozta meg eddig az AlphaFold
Amint két évvel ezelőtti cikkünkben összefoglaltuk, az AlphaFold2 fehérjekutató algoritmus volt az első, amely megbízhatóan volt képes az aminosav-szekvenciából fehérjék háromdimenziós szerkezetének megállapítására. Ennek alapját a fehérjék aminosavsorrendjének meghatározása adja, ami egy szekvenálásnak nevezett eljárással történik. Az algoritmus ebből a szekvenciából indul ki, és információt nyer még azonos evolúciós eredetű fehérjék egymáshoz illesztett aminosavsorrendjéből is – azaz a feltekeredett szerkezet megállapításához evolúciós információt is használ.
A DeepMind és az Isomorphic Labs májusban mutatta be az AlphaFold2 utódját, az AlphaFold3-at, amely már a DNS és RNS háromdimenziós szerkezetének meghatározására is képes. „Az AlphaFold3 bejelentése fontos mérföldkő” – mondta akkor az algoritmus bemutatását megelőző sajtótájékoztatón Hassabis. Az új fehérjekutató algoritmus pontosabb is az elődjénél, ami fontos lépés ahhoz, hogy praktikusan használható legyen a gyógyszertervezés során. Emellett az algoritmus architektúrája kevésbé alapszik az említett evolúciós információ feltárásán.
Az AlphaFold3-at az Isomorphic Labs AI-kutatója, Max Jaderberg akkor áttörőnek nevezte, és már most ígéretesnek tűnik a gyógyszerhatóanyagok felfedezésének felgyorsításábanó és az egész hatóanyag-kutatási folyamat átalakításában.
Holnap az irodalmi díj bejelentésével folytatódik a Nobel-hét
Az előző években egzotikus témákat díjaztak a kémiai kategóriában: tavaly a kvantumpontok felfedezéséért járt az elismerés, 2022-ben a klikk-kémia úttörői nyerték a díjat, 2021-ben pedig az úgynevezett aszimmetrikus organokatalízis kifejlesztéséért vehették át díjukat a győztesek.
A 2024-es Nobel-díjak bejelentése csütörtökön az irodalmi, majd pénteken a békedíjjal folytatódik, míg október 14-én hétfőn kihirdetik az 1969-ben alapított közgazdasági Nobel-emlékdíj győztesét vagy győzteseit is.
A Nobel-díjat Alfred Nobel svéd kémikus és feltaláló hozta létre, aki hagyatékát az emberiségnek legnagyobb hasznot hozó kutatók elismerésére ajánlotta fel – az első díjakat halála után hat évvel, 1901-ben adták át. Az elismeréseket hivatalosan idén is december 10-én, Nobel halálának napján adják majd át Stockholmban, amikor a kitüntetett kutatók a Nobel-díjhoz járó érem és diploma mellett 11 millió svéd koronát (nagyjából 390 millió forintot) is kapnak.
Kapcsolódó cikkek